Àcid ribonucleic
| Part d'una sèrie sobre |
| Genètica |
|---|
 |
|
Conceptes fonamentals |
|
Medicina de precisió |
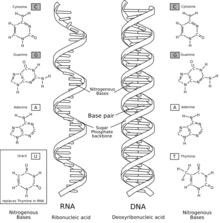
L'àcid ribonucleic (ARN, conegut igualment per la sigla anglesa RNA) és una molècula polimèrica que té un paper clau en la codificació, la descodificació, la regulació i l'empalmament dels gens. L'ARN i l'ADN són àcids nucleics, que constitueixen els quatre tipus de macromolècules essencials per a la vida juntament amb els lípids, les proteïnes i els glúcids. Tant l'ADN com l'ARN consisteixen en una cadena de nucleòtids, el primer en forma de doble cadena helicoidal i el segon en forma d'una sola cadena que es plega sobre si mateixa.
Consisteix en una llarga cadena de nucleòtids units entre ells. Cada nucleòtid conté una base nitrogenada, una ribosa (sucre) i un grup fosfat. L'ARN és molt similar estructuralment a l'ADN, però té petites diferències pel que fa a l'estructura: en la cèl·lula, l'ARN es presenta en forma de cadena simple; mentre que l'ADN hi és present en forma de doble cadena; els nucleòtids d'ADN contenen desoxiribosa (un tipus de ribosa que té un àtom d'oxigen menys); i l'ARN té la base nitrogenada uracil en lloc de la timina, que és present a l'ADN.

L'ARN es transcriu a partir de l'ADN per enzims anomenats ARN polimerases i és un procés regulat també per altres enzims. L'ARN té una funció central en la síntesi de proteïnes. En aquest cas, un tipus d'ARN, anomenat ARN missatger, porta informació des de l'ADN cap als ribosomes. Aquests ribosomes estan formats de proteïnes i ARN ribosomal, que s'assemblen per formar una molècula que actua com una màquina que pot llegir els ARN missatgers i traduir la informació que porten per formar proteïnes. Hi ha altres ARN que tenen funcions diferents – en particular la de regular quins gens s'expressen, però també quins genomes dels virus s'expressen.
Estructura

Cada nucleòtid de l'ARN conté un sucre del tipus ribosa, amb carbonis numerats de l'1' al 5'. Cada ribosa està unida, en la posició 1', a una base nitrogenada, generalment adenina (A), citosina (C), guanina (G) o uracil (U). L'adenina i la guanina són purines, mentre que la citosina i l'uracil són pirimidines. Un grup fosfat és el darrer element de l'ARN, que s'uneix a aquesta mateixa ribosa per la posició 3' i, a la ribosa següent, per la posició 5', de manera que serveix d'unió de la cadena. Els grups fosfat tenen càrrega negativa a pH fisiològic, convertint l'ARN en una molècula carregada (polianió). Les bases poden formar ponts d'hidrogen entre citosina i guanina, entre adenina i uracil i entre guanina i uracil.[1] De totes maneres, són possibles altres interaccions, com la de dues adenines en un bucle,[2] o el GNRA tetraloòping, que té un parell de bases de guanina-adenina.[3]

Un tret important de l'ARN que el distingeix de l'ADN és la presència d'un grup hidroxil a la posició 2' de la ribosa. La presència d'aquest grup funcional és la causa que l'hèlix adopti la geometria A (hèlix α) més sovint que la forma B (hèlix β), més habitual en l'ADN.[4] Una segona conseqüència de la presència del 2'-hidroxil és que, en zones conformalment flexibles de l'ARN (és a dir, no involucrades en la formació d'una doble hèlix), pot assaltar químicament el pont fosfodièster adjacent per tal de clavar-se al pilar.[5]

L'ARN és transcrit només amb quatre bases (adenina, guanina, citosina i uracil),[6] però hi ha nombroses bases i sucres modificats a l'ARN madur. La pseudouridina (Ψ), en la qual la unió entre l'uracil i la ribosa és modificada d'un pont C-N a un pont C-C, i la ribotimidina (T) es localitzen en diversos llocs (el més important, la volta TΨC de l'ARNt).[7] Una altra base modificada notable és la inosina (I). La inosina té un paper clau en el codi genètic.[8] Hi ha prop de 100 altres nucleòsids espontàniament modificats,[9] dels quals la pseudouridina i els nucleòsids amb 2'-O-metilribosa en són els més comuns.[10] Els papers específics de moltes d'aquestes modificacions de l'ARN no estan encara del tot esbrinats. De totes maneres, cal remarcar que en l'ARN ribosòmic, moltes d'aquestes modificacions post-transcripcionals ocorren en regions altament funcionals, com ara el centre i la subunitat interfàsica de la peptidiltransferasa, suggerint un paper important en la funcionalitat normal.[11]
La forma funcional d'una molècula d'ARN monocatenària, igual que les proteïnes, requereix sovint una estructura terciària específica. La bastida per a aquesta estructura és proporcionada pels elements de l'estructura secundària, és a dir, els ponts d'hidrogen de dins la molècula. Això dirigeix diversos dominis reconoscibles, fulles plegades, llaços, bucles i altres formes.[1] Com que l'ARN està carregat, es necessiten els ions metàl·lics com el Mg2+ per estabilitzar l'estructura secundària.[12]
Classificació
Els àcids ribonucleics se solen tipificar segons la seva funció biològica en ARN missatger (ARNm o mRNA), ARN de transferència (ARNt o tRNA), ARN ribosòmic (ARNr o rRNA). Tanmateix es poden diferenciar per la seva estructura en ARN bicatenari o ARN de cadena doble complementària que consta de dos filaments (en els reovirus) i ARN monocatenari.
Comparació amb l'ADN

L'ARN i l'ADN són àcids nucleics, però tenen tres diferències importants. Primer de tot, l'ADN és de doble cadena, mentre que l'ARN és de cadena simple i en la majoria de funcions que ha de fer té una cadena més curta de nucleòtids. Segonament, mentre que l'ADN conté una desoxiribosa, l'ARN conté una ribosa (no té grup hidroxil enganxat a l'anell que forma la pentosa en la posició 2' de l'ADN). Aquests grups hidroxil fan que l'ARN sigui menys estable que l'ADN perquè és més fàcil d'hidrolitzar. Com a tercer, la base complementària de l'adenina no és la timina, com en el cas de l'ADN, sinó que és l'uracil, que és una forma no metilada de la timina.[13]
Igual que l'ADN, la majoria d'ARN actius, inclòs l'ARNm, ARNt, ARNr, ARNsn i altres ARN no codificants, contenen seqüències complementàries d'ells mateixos, que permeten que diverses parts de la cadena s'uneixin i formin dobles hèlixs. Diverses anàlisis d'aquest ARN han revelat que tenen una estructura molt complexa. Al contrari de l'ADN, les seves estructures no consisteixen en dobles hèlixs, sinó que són col·leccions d'hèlixs curtes empaquetades en estructures semblants a proteïnes. Per aquesta raó, l'ARN pot catalitzar reaccions com ho fa un enzim.[14] Per exemple, la determinació de l'estructura del ribosoma (un enzim que catalitza la formació d'un enllaç peptídic) va revelar que és aquest lloc d'activitat es compon solament d'ARN.[15]
Síntesi
La síntesi d'ARN està catalitzada normalment per un enizm (l'ARN polimerasa), que fa servir l'ADN com a plantilla, en un procés que s'anomena transcripció. El començament de la transcripció es fa amb la unió de l'enzim amb una seqüència promotora (que normalment es troba al començament del gen). L'ADN de doble hèlix es descaragola gràcies a l'acció de l'helicasa. Llavors, l'enzim avança al llarg de la cadena en sentit 3' cap a 5'. La seqüència d'ADN també dicta quan acaba la síntesi d'ARN.[16]
L'ARN sovint es veu modificat per enzims després de la transcripció. Per exemple, s'afegeix una cua de poliA i un cap a l'extrem 5' i els introns són eliminats per l'espliceosoma.
També hi ha un gran nombre d'ARN polimerases dependents de l'ARN, que utilitzen l'ARN com a plantilla per la síntesi d'ARN. Per exemple, un gran nombre d'ARN de virus (com el virus de la poliomielitis) utilitzen aquest tipus d'enzim per replicar el seu material genètic.[17] A més a més, els d'ARN polimerases dependents de l'ARN són part de la interferència de l'ARN en molts organismes[18]
Tipus
L'ARN missatger és l'ARN que du informació des de l'ADN fins al ribosoma, el lloc on es produeix la síntesi de proteïnes (traducció) a la cèl·lula.[19] La seqüència codificada d'ARN missatger determina la seqüència d'aminoàcids en la proteïna que s'està sintetitzant. Tot i això, molts ARN no codifiquen proteïna (vora un 97% de la cadena no és transcriptora, en el cas de cèl·lules eucariotes)[20][21][22][23]).
Aquests fragments que no codifiquen (anomenats ARN no codificants o ncARN) poden ser codificats pels seus propis gens (gens ARN) però també poden derivar dels introns dels ARN missatgers.[24] L'exemple més important d'ARN no codificant és l'ARN de transferència (ARNt) i l'ARN ribosòmic (ARNr). Tots dos estan involucrats en la regulació de gens, el processament d'ARN i altres funcions.[13] Certs ARN poden catalitzar reaccions químiques com per exemple tallar o unir altres ARN moleculars,[25] o la catàlisi d'enllaços peptídics dels ribosomes,[15] anomenats ribozimes.

Traducció
L'ARN missatger (ARNm) porta informació sobre la proteïna als ribosomes, les fàbriques de síntesi de proteïnes a la cèl·lula. Es codifica de manera que cada tres nucleòtids (un codó) es forma un aminoàcid. En les cèl·lules eucariotes, un precursor d'ARN missatger (el pre-ARN missatger) es transcriu a partir de l'ADN, llavors es processa cap a ARN madur. Aquest últim pas suposa l'eliminació d'introns – les regions no codificants del pre-ARNm. Llavors, aquest ARN s'exporta del nucli al citoplasma, on s'uneix als ribosomes i es tradueix la proteïna corresponent amb l'ajut de l'ARNt. En les cèl·lules procariotes, que no tenen ni nucli ni citoplasma, l'ARN missatger pot unir-se als ribosomes mentre està sent transcrit a partir de l'ADN. Després d'un cert temps, el missatge es degrada amb l'ajut de les ribonucleases.[19]
L'ARN de transferència (ARNt) és un ARN petit, format de 80 nucleòtids que transfereix un aminoàcid específic a un polipèptid creixent i això ho fa a la part del ribosoma on hi ha la síntesi de proteïna durant la traducció. També té lloc per adjuntar els aminoàcids i una regió anticodó per reconèixer el codó, que s'uneix a una seqüència específica de la cadena d'ARN mitjançant un pont d'hidrogen.[24]
L'ARN ribosomal (ARNr) és el component catalític dels ribosomes. Els ribosomes eucariotes conenen 4 tipus diferents de molècules d'ARNr: 18S, 5.8S, 28S i 5S ARNr. Tres d'elles són sintetitzades al nucli cel·lular i una se sintetitza en un altre lloc. En el citoplasma, l'ARN ribosòmic i la proteïna es combinen per formar una nucleoproteïna anomenada ribosoma. El ribosoma s'uneix a l'ARNm i du a terme la síntesi de proteïnes. Molts ribosomes poden unir-se a un ARN lliure en qualsevol moment.[19] L'ARNr és extremadament abundant i representa un 80% de l'ARN trobat en el citoplasma d'una cèl·lula eucariota.[26]
L'ARN de transferència-missatger (tmARN) es troba en diversos bacteris i plastidis. Aquest etiqueta proteïnes codificades per l'ARN missatger que no tenen codons de terminació a causa de la degradació.[27]
ARN de regulació
Diversos tipus d'ARN poden provocar una mala regulació de l'expressió dels gens mitjançant la seva complementarietat amb una part de l'ARNm o un gen d'ADN. Els microARN (miARN; 21-22 nt) es poden trobar en eucariotes i actuen mitjançant una interferència d'ARN (ARNi), en la qual un complex de miARn i els enzims poden trencar l'ARNm que li és complementari, poden bloquejar l'ARNm i que no es pugui traduir o bé accelerar la seva degradació.[28][29] A més a més, l'ARN d'interferència petit (siARN; 20-25nt) normalment és produït per un trencament de l'ARN viral.[30][31] Aquest siARN pot interferir en l'ARN d'una manera semblant al miARN. Alguns miARN i siARN poden causar la metilació d'alguns gens, o bé el decreixement o creixement de transcripció d'aquests gens. [32][33][34]
Els animals tenen ARN Piwi-d'interacció (piARN; 29-30 nt), que estan actius a les cèl·lules i es pensa que són un mecanisme de defensa contra els transposons i tenen una funció important en la gametogènesis.[35][36] Tots els procariotes tenen ARN CRISPR, un sistema de regulació semblant a la interferència d'ARN.[37] Els Antisense ARN estan estesos; la majoria provoquen una mala regulació dels gens, però pocs d'ells són activadors de la transcripció.[38] L'antisense ARN pot actuar unint-se a un ARNmissatger, formant un ARN de doble cadena que és degradat per enzims.[39] Hi ha molts ARN llargs no-codificants que regulen els gens en els eucariotes,[40] com per exemple ARN Xist, que cobreix un cromosoma X en els mamífers femelles i l'inactiva.[41]
Un ARN pot contenir elements reguladors com per exemple “riboswitches”, en la regió que no transcriu 5' o en la regió que no transcriu 3'; aquests elements cis-reguladors regulen l'activitat de l'ARNm.[42] Les regions que no han estat traduïdes també poden tenir elements que regulin altres gens.[43]
ARN processador
Alguns ARN modifiquen altres ARN. Els introns són desenganxats dels pre-ARNm pels espliceosomes, que contenen diversos ARN nuclears petits;[13] o bé podem trobar ribozimes que desenganxin aquests introns.[44]
També es pot modificar l'ARN mitjançant l'alteració dels seus nucleòtids; aquesta alteració és produïda per altres nucleòtids (A, C, G i U). En els eucariotes, és l'ARN nuclear petit (snoARN; 60-300 nt)[24] el que dirigeix la modificació de l'ARN, que es pot trobar al nucli cel·lular i als cossos de Cajal. El snoARN s'associa amb enzims i llavors porta a terme la modificació del nuclèotid. Els ARNr i ARNt es modifiquen de manera important, però les snARN i ARNm també poden patir modificacions.[45][46]
Genomes d'ARN
Igual que l'ADN, l'ARN pot dur informació genètica. Els virus d'ARN tenen genomes compostos d'ARN, i una varietat de proteïnes que són codificades per aquest genoma. Algunes d'aquestes proteïnes són les encarregades de replicar el genoma viral, mentre que altres proteïnes protegeixen el genoma quan la partícula vírica es mou cap a una altra cèl·lula. Els virions són un altre grup de patògens però només consten d'ARN, i no codifiquen cap proteïna i són replicades per una polimerasa de la cèl·lula que ha parasitat.[47]

Retrotranscripció
Els retrovirus repliquen els seus gens per transcripció inversa. L'ADN es copia de l'ARN, i aquestes còpies d'ADN es transcriuen cap a ARN. Els retrotransposons se separen copiant l'ADN i l'ARN d'altres còpies,[48] i la telomerasa conté un ARN que és usat com a base per construir la part final dels cromosomes de les cèl·lules eucariotes.[49]
ARN de cadena doble
L'ARN de doble cadena (dsARNde l'anglès Double Stranded RNA) és un ARN amb dues cadenes complementàries, similar a l'ADN que hi ha a les cèl·lules. Els dsARN formen el material genètic d'alguns virus (virus ARN bicatenari). L'ARN de doble cadena com per exemple el dels virus o siARN pot provocar la interferència d'ARN en el cas de cèl·lules eucariotes, així com provocar respostes, també com a interferó en els vertebrats.[50][51][52]
Descobriment
Els àcids nucleics foren descoberts l'any 1868 per Friedrich Miescher, que va anomenar “nucleina” a aquest material, perquè es trobava al nucli.[53] Més tard es va descobrir en les cèl·lules procariotes, que no tenen nucli però també contenen àcids nucleics. La funció de l'ARN en la síntesi de proteïnes es va començar a descobrir l'any 1939.[54] Severo Ochoa va guanyar el Premi Nobel de Medicina l'any 1959 després de descobrir com se sintetitzava l'ARN.[55] Robert W. Holley, l'any 1965,[56] va guanyar el Premi Nobel de Medicina l'any 1968 per descobrir la seqüència de 77 nucleòtids de l'ARNt.
L'any 1967, Carl Woese va dir que l'ARN pot ser catalitzat i va proposar que les primeres formes de vida es basaven en l'ARN per portar informació genètica per catalitzar reaccions bioquímiques – un món d'ARN.[57][58] L'any 1976, Walter Fiers i el seu equip van determinar la primera seqüència d'un gen d'ARN d'un virus, del bacteriophage MS2.[59]
L'any 1990 es va descobrir que alguns gens poden inactivar gens de la mateixa planta, fet que ara es coneix com a ARN d'interferència.[60][61] Al mateix temps es va descobrir que els ARN llargs de 22 nt, que ara s'anomenen microARN, tenien una funció en el desenvolupament de Caenorhabditis elegans.[62] El descobriment de la regulació de gens que es du a terme mitjançant l'ARN ha permès fer intents en el desenvolupament de drogues compostes d'ARN, com per exemple siARN, per tal d'inactivar gens.[63]
Flux d'informació genètica
En la majoria dels organismes, el cicle d'informació genètica comença a l'ADN i es bifurca en dos possibles camins: la replicació (que òbviament produeix un nou ADN idèntic) i la transcripció, que produeix l'ARN. Aquest ARN pot ésser convertit en proteïnes mitjançant un procés anomenat traducció.
L'ARN com a eina terapèutica
L'ARN te diverses aplicacions terapèutiques, com ara els vaccins d'ARN, l'ARN d'interferència, l'ARN antisense o els sensors d'ARN programables.
Referències
- ↑ 1,0 1,1 Mathews DH, Disney MD, Childs JL, Schroeder SJ, Zuker M, Turner DH «Incorporating chemical modification constraints into a dynamic programming algorithm for prediction of RNA secondary structure». Proc. Natl. Acad. Sci. USA, 101, 19, 2004, pàg. 7287–92. DOI: 10.1073/pnas.0401799101. PMID: 15123812.
- ↑ Barciszewski J, Frederic B, Clark C. RNA biochemistry and biotechnology. Springer, 1999, p. 73–87. ISBN 0792358627. OCLC 52403776.
- ↑ Lee JC, Gutell RR «Diversity of base-pair conformations and their occurrence in rRNA structure and RNA structural motifs». J. Mol. Biol., 344, 5, 2004, pàg. 1225–49. DOI: 10.1016/j.jmb.2004.09.072. PMID: 15561141.
- ↑ Salazar M, Fedoroff OY, Miller JM, Ribeiro NS, Reid BR «The DNA strand in DNAoRNA hybrid duplexes is neither B-form nor A-form in solution». Biochemistry, 32, 16, 1992, pàg. 4207–15. DOI: 10.1021/bi00067a007. PMID: 7682844.
- ↑ Mikkola S, Nurmi K, Yousefi-Salakdeh E, Strömberg R, Lönnberg H «The mechanism of the metal ion promoted cleavage of RNA phosphodiester bonds involves a general acid catalysis by the metal aquo ion on the departure of the leaving group». Perkin transactions 2, 1999, pàg. 1619–26. DOI: 10.1039/a903691a.
- ↑ Jankowski JAZ, Polak JM. Clinical gene analysis and manipulation: Tools, techniques and troubleshooting. Cambridge University Press, 1996, p. 14. ISBN 0521478960. OCLC 33838261.
- ↑ Yu Q, Morrow CD «Identification of critical elements in the tRNA acceptor stem and TΨC loop necessary for human immunodeficiency virus type 1 infectivity». J Virol., 75, 10, 2001, pàg. 4902–6. DOI: 10.1128/JVI.75.10.4902-4906.2001. PMID: 11312362.
- ↑ Elliott MS, Trewyn RW «Inosine biosynthesis in transfer RNA by an enzymatic insertion of hypoxanthine». J. Biol. Chem., 259, 4, 1983, pàg. 2407–10. PMID: 6365911.
- ↑ Söll D, RajBhandary U. TRNA: Structure, biosynthesis, and function. ASM Press, 1995, p. 165. ISBN 155581073X. OCLC 183036381 30663724.
- ↑ Kiss T «Small nucleolar RNA-guided post-transcriptional modification of cellular RNAs». The EMBO Journal, 20, 2001, pàg. 3617–22. DOI: 10.1093/emboj/20.14.3617. PMID: 11447102.
- ↑ King TH, Liu B, McCully RR, Fournier MJ «Ribosome structure and activity are altered in cells lacking snoRNPs that form pseudouridines in the peptidyl transferase center». Molecular Cell, 11, 2, 2002, pàg. 425–35. DOI: 10.1016/S1097-2765(03)00040-6. PMID: 12620230.
- ↑ Tan ZJ, Chen SJ «Salt dependence of nucleic acid hairpin stability». Biophys. J., 95, 2, 2008, pàg. 738–52. DOI: 10.1529/biophysj.108.131524. PMID: 18424500.
- ↑ 13,0 13,1 13,2 Berg JM, Tymoczko JL, Stryer L. Biochemistry. 5a edició. WH Freeman and Company, 2002, p. 118–19, 781–808. ISBN 0-7167-4684-0. OCLC 179705944 48055706 59502128.
- ↑ Higgs PG «RNA secondary structure: physical and computational aspects». Quarterly Reviews of Biophysics, 33, 2000, pàg. 199–253. DOI: 10.1017/S0033583500003620. PMID: 11191843.
- ↑ 15,0 15,1 Nissen P, Hansen J, Ban N, Moore PB, Steitz TA «The structural basis of ribosome activity in peptide bond synthesis». Science, 289, 5481, 2000, pàg. 920–30. DOI: 10.1126/science.289.5481.920. PMID: 10937990.
- ↑ Nudler E, Gottesman ME «Transcription termination and anti-termination in E. coli». Genes to Cells, 7, 2002, pàg. 755–68. DOI: 10.1046/j.1365-2443.2002.00563.x. PMID: 12167155.
- ↑ Jeffrey L Hansen, Alexander M Long, Steve C Schultz «Structure of the RNA-dependent RNA polymerase of poliovirus». Structure, 5, 8, 1997, pàg. 1109–22. DOI: 10.1016/S0969-2126(97)00261-X. PMID: 9309225.
- ↑ Ahlquist P «RNA-Dependent RNA Polymerases, Viruses, and RNA Silencing». Science, 296, 5571, 2002, pàg. 1270–73. DOI: 10.1126/science.1069132. PMID: 12016304.
- ↑ 19,0 19,1 19,2 Cooper GC, Hausman RE. The Cell: A Molecular Approach. 3a ed.. Sinauer, 2004, p. 261–76, 297, 339–44. ISBN 0-87893-214-3. OCLC 174924833 52121379 52359301 56050609.
- ↑ Mattick JS, Gagen MJ «The evolution of controlled multitasked gene networks: the role of introns and other noncoding RNAs in the development of complex organisms». Mol. Biol. Evol., 18, 9, 01-09-2001, pàg. 1611–30. PMID: 11504843.
- ↑ Mattick, J.S. (2001). “Noncoding RNAs: the architects of eukaryotic complexity”. EMBO Reports, 2(11), 986-991. «emboreports.npgjournals.com». Arxivat de l'original el 2005-12-27. [Consulta: 16 desembre 2009].
- ↑ Mattick, J.S. (2003). “Challenging the dogma: The hidden layer of non-protein-coding RNAs on complex organisms” Bioessays. 25, 930-939. «PDF».
- ↑ Mattick, J.S. (2004). “The hidden genetic program of complex organisms” Scientific American. 291(4), 30-37. «Enllaç».
- ↑ 24,0 24,1 24,2 Wirta W. Mining the transcriptome – methods and applications. Stockholm: School of Biotechnology, Royal Institute of Technology, 2006. ISBN 91-7178-436-5. OCLC 185406288.
- ↑ Rossi, JJ «Ribozyme diagnostics comes of age». Chemistry & Biology, 11, 7, 2004, pàg. 894–95. DOI: 10.1016/j.chembiol.2004.07.002. PMID: 15271347.
- ↑ Kampers T, Friedhoff P, Biernat J, Mandelkow E-M, Mandelkow E «RNA stimulates aggregation of microtubule-associated protein tau into Alzheimer-like paired helical filaments». FEBS Letters, 399, 3, 1996, pàg. 104D. DOI: 10.1016/S0014-5793(96)01386-5. PMID: 8985176.
- ↑ Gueneau de Novoa P, Williams KP «The tmRNA website: reductive evolution of tmRNA in plastids and other endosymbionts». Nucleic Acids Res., 32, Database issue, 2004, pàg. D104–8. DOI: 10.1093/nar/gkh102. PMID: 14681369.
- ↑ Wu L, Belasco JG «Let me count the ways: mechanisms of gene regulation by miRNAs and siRNAs». Mol. Cell, 29, 1, gener 2008, pàg. 1–7. DOI: 10.1016/j.molcel.2007.12.010. PMID: 18206964.
- ↑ Matzke MA, Matzke AJM «Planting the seeds of a new paradigm». PLoS Biology, 2, 5, 2004, pàg. e133. DOI: 10.1371/journal.pbio.0020133. PMID: 15138502.
- ↑ Vazquez F, Vaucheret H, Rajagopalan R, Lepers C, Gasciolli V, Mallory AC, Hilbert J, Bartel DP, Crété P «Endogenous trans-acting siRNAs regulate the accumulation of Arabidopsis mRNAs». Molecular Cell, 16, 1, 2004, pàg. 69–79. DOI: 10.1016/j.molcel.2004.09.028. PMID: 15469823.
- ↑ Watanabe T, Totoki Y, Toyoda A, et al. «Endogenous siRNAs from naturally formed dsRNAs regulate transcripts in mouse oocytes». Nature, 453, 7194, maig 2008, pàg. 539–43. DOI: 10.1038/nature06908. PMID: 18404146.
- ↑ Sontheimer EJ, Carthew RW «Silence from within: endogenous siRNAs and miRNAs». Cell, 122, 1, juliol 2005, pàg. 9–12. DOI: 10.1016/j.cell.2005.06.030. PMID: 16009127.
- ↑ Doran G «RNAi – Is one suffix sufficient?». Journal of RNAi and Gene Silencing, 3, 1, 2007, pàg. 217–19. Arxivat de l'original el 2007-07-16 [Consulta: 16 desembre 2009].
- ↑ Pushparaj PN, Aarthi JJ, Kumar SD, Manikandan J «RNAi and RNAa - The Yin and Yang of RNAome». Bioinformation, 2, 6, 2008, pàg. 235–7. PMC: 2258431. PMID: 18317570.
- ↑ Horwich MD, Li C Matranga C, Vagin V, Farley G, Wang P, Zamore PD «The Drosophila RNA methyltransferase, DmHen1, modifies germline piRNAs and single-stranded siRNAs in RISC». Current Biology, 17, 2007, pàg. 1265–72. DOI: 10.1016/j.cub.2007.06.030. PMID: 17604629.
- ↑ Girard A, Sachidanandam R, Hannon GJ, Carmell MA «A germline-specific class of small RNAs binds mammalian Piwi proteins». Nature, 442, 2006, pàg. 199–202. DOI: 10.1038/nature04917. PMID: 16751776.
- ↑ Makarova KS, Grishin NV, Shabalina SA, Wolf YI, Koonin EV «A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action». Biol. Direct, 1, 2006, pàg. 7. Arxivat de l'original el 2013-05-10. DOI: 10.1186/1745-6150-1-7. PMID: 16545108 [Consulta: 16 desembre 2009].
- ↑ Wagner EG, Altuvia S, Romby P «Antisense RNAs in bacteria and their genetic elements». Adv Genet., 46, 2002, pàg. 361–98. DOI: 10.1016/S0065-2660(02)46013-0. PMID: 11931231.
- ↑ Gilbert SF. Developmental Biology. 7a ed.. Sinauer, 2003, p. 101–3. ISBN 0878932585. OCLC 154656422 154663147 174530692 177000492 177316159 51544170 54743254 59197768 61404850 66754122.
- ↑ Amaral PP, Mattick JS «Noncoding RNA in development». Mammalian genome : official journal of the International Mammalian Genome Society, 19, 7-8, octubre 2008, pàg. 454. DOI: 10.1007/s00335-008-9136-7. PMID: 18839252.
- ↑ Heard E, Mongelard F, Arnaud D, Chureau C, Vourc'h C, Avner P «Human XIST yeast artificial chromosome transgenes show partial X inactivation center function in mouse embryonic stem cells». Proc. Natl. Acad. Sci. USA, 96, 12, 1999, pàg. 6841–46. DOI: 10.1073/pnas.96.12.6841. PMID: 10359800.
- ↑ Batey RT «Structures of regulatory elements in mRNAs». Curr. Opin. Struct. Biol., 16, 3, 2006, pàg. 299–306. DOI: 10.1016/j.sbi.2006.05.001. PMID: 16707260.
- ↑ Scotto L, Assoian RK «A GC-rich domain with bifunctional effects on mRNA and protein levels: implications for control of transforming growth factor beta 1 expression». Mol. Cell. Biol., 13, 6, juny 1993, pàg. 3588–97. PMC: 359828. PMID: 8497272.
- ↑ Steitz TA, Steitz JA «A general two-metal-ion mechanism for catalytic RNA». Proc. Natl. Acad. Sci. U.S.A., 90, 14, 1993, pàg. 6498–502. DOI: 10.1073/pnas.90.14.6498. PMID: 8341661.
- ↑ Xie J, Zhang M, Zhou T, Hua X, Tang L, Wu W «Sno/scaRNAbase: a curated database for small nucleolar RNAs and cajal body-specific RNAs». Nucleic Acids Res., 35, Database issue, 2007, pàg. D183–7. DOI: 10.1093/nar/gkl873. PMID: 17099227.
- ↑ Omer AD, Ziesche S, Decatur WA, Fournier MJ, Dennis PP «RNA-modifying machines in archaea». Molecular Microbiology, 48, 3, 2003, pàg. 617–29. DOI: 10.1046/j.1365-2958.2003.03483.x. PMID: 12694609.
- ↑ Daròs JA, Elena SF, Flores R «Viroids: an Ariadne's thread into the RNA labyrinth». EMBO Rep., 7, 6, 2006, pàg. 593–8. DOI: 10.1038/sj.embor.7400706. PMID: 16741503.
- ↑ Kalendar R, Vicient CM, Peleg O, Anamthawat-Jonsson K, Bolshoy A, Schulman AH «Large retrotransposon derivatives: abundant, conserved but nonautonomous retroelements of barley and related genomes». Genetics, 166, 3, 2004, pàg. D339. DOI: 10.1534/genetics.166.3.1437. PMID: 15082561.
- ↑ Podlevsky JD, Bley CJ, Omana RV, Qi X, Chen JJ «The telomerase database». Nucleic Acids Res., 36, Database issue, 2008, pàg. D339–43. DOI: 10.1093/nar/gkm700. PMID: 18073191.
- ↑ Blevins T et al. «Four plant Dicers mediate viral small RNA biogenesis and DNA virus induced silencing». Nucleic Acids Res, 34, 21, 2006, pàg. 6233–46. DOI: 10.1093/nar/gkl886. PMID: 17090584.
- ↑ Jana S, Chakraborty C, Nandi S, Deb JK «RNA interference: potential therapeutic targets». Appl. Microbiol. Biotechnol., 65, 6, 2004, pàg. 649–57. DOI: 10.1007/s00253-004-1732-1. PMID: 15372214.
- ↑ Schultz U, Kaspers B, Staeheli P «The interferon system of non-mammalian vertebrates». Dev. Comp. Immunol., 28, 5, 2004, pàg. 499–508. DOI: 10.1016/j.dci.2003.09.009. PMID: 15062646.
- ↑ Dahm R «Friedrich Miescher and the discovery of DNA». Developmental Biology, 278, 2, 2005, pàg. 274–88. DOI: 10.1016/j.ydbio.2004.11.028. PMID: 15680349.
- ↑ Caspersson T, Schultz J «Pentose nucleotides in the cytoplasm of growing tissues». Nature, 143, 1939, pàg. 602–3. DOI: 10.1038/143602c0.
- ↑ Ochoa S. «Enzymatic synthesis of ribonucleic acid». Nobel Lecture, 1959.
- ↑ Holley RW et al. «Structure of a ribonucleic acid». Science, 147, 1664, 1965, pàg. 1462–65. DOI: 10.1126/science.147.3664.1462. PMID: 14263761.
- ↑ Siebert S. «Common sequence structure properties and stable regions in RNA secondary structures». Dissertation, Albert-Ludwigs-Universität, Freiburg im Breisgau p. 1, 2006. Arxivat de l'original el 2012-03-09. [Consulta: 16 desembre 2009].
- ↑ Szathmáry E «The origin of the genetic code: amino acids as cofactors in an RNA world». Trends Genet., 15, 6, 1999, pàg. 223–9. DOI: 10.1016/S0168-9525(99)01730-8. PMID: 10354582.
- ↑ Fiers W et al. «Complete nucleotide-sequence of bacteriophage MS2-RNA: primary and secondary structure of replicase gene». Nature, 260, 5551, 1976, pàg. 500–7. DOI: 10.1038/260500a0. PMID: 1264203.
- ↑ Napoli C, Lemieux C, Jorgensen R «Introduction of a chimeric chalcone synthase gene into petunia results in reversible co-suppression of homologous genes in trans». Plant Cell, 2, 4, 1990, pàg. 279–89. DOI: 10.1105/tpc.2.4.279. PMID: 12354959.
- ↑ Dafny-Yelin M, Chung SM, Frankman EL, Tzfira T «pSAT RNA interference vectors: a modular series for multiple gene down-regulation in plants». Plant Physiol., 145, 4, desembre 2007, pàg. 1272–81. DOI: 10.1104/pp.107.106062. PMC: 2151715. PMID: 17766396.
- ↑ Ruvkun G «Glimpses of a tiny RNA world». Science, 294, 5543, 2001, pàg. 797–99. DOI: 10.1126/science.1066315. PMID: 11679654.
- ↑ Fichou Y, Férec C «The potential of oligonucleotides for therapeutic applications». Trends in Biotechnology, 24, 12, 2006, pàg. 563–70. DOI: 10.1016/j.tibtech.2006.10.003. PMID: 17045686.
Vegeu també
| Principals famíles bioquímiques | ||
| Àcids nucleics | Alcaloides | Aminoàcids | Carbohidrats | Carotenoides | Cofactors enzimàtics | Esteroides | Flavonoides | Glicòsids | Lípids | Pèptids | Policètids | Tetrapirrols | Terpens | ||
| Anàlegs d'àcids nucleics: | Tipus d'àcids nucleics | Anàlegs d'àcids nucleics : |
| Bases nitrogenades: | Adenina | Timina | Uracil | Guanina | Citosina | Purina | Pirimidina | |
|---|---|---|
| Nucleòsids: | Adenosina | Uridina | Guanosina | Citidina | Desoxiadenosina | Timidina | Desoxiguanosina | Desoxicitidina | |
| Nucleòtids: | AMP | UMP | GMP | CMP | ADP | UDP | GDP | CDP | ATP | UTP | GTP | CTP | AMPc | GMPc | ADPRc | |
| Desoxinucleòtids: | dAMP | TMP | dGMP | dCMP | dADP | TDP | dGDP | dCDP | dATP | TTP | dGTP | dCTP | |
| Àcids ribonucleics: | ARNm | ARNt | ARNr | ARNn | ARNnc | ARNmi | |
| Àcids desoxiribonucleics: | ADMmt | ADNc | |
| Anàlegs d'àcids nucleics: | AGN | APN | ATN | Morfolí | ARNin | |
| Seqüències: | Plasmidi | Còsmid | CAB | CAH | Cromosoma | Oligonucleòtid | |
