Haplotipo
Un haplotipo (del gr. ἁπλόος, haplóos, ‘único, simple’) en genética es una combinación de alelos de diferentes loci de un cromosoma que son transmitidos juntos. Un haplotipo puede ser un locus, varios loci, o un cromosoma entero dependiendo del número de eventos de recombinación que han ocurrido entre un conjunto dado de loci.
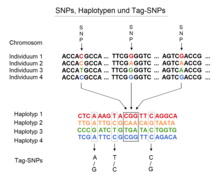
En un segundo significado, un haplotipo es un conjunto de polimorfismo de un solo nucleótido (SNPs) en un cromosoma particular que están estadísticamente asociados.
Como se ha indicado anteriormente, un haplotipo es la constitución alélica de múltiples loci para un mismo cromosoma. Dada la alta variabilidad alélica en el genoma humano, la probabilidad de que dos individuos no relacionados presenten un mismo haplotipo, es prácticamente nula. Es por esto que el estudio de haplotipos se ha convertido en una herramienta útil en la determinación de relación génica entre individuos, y por lo tanto en el estudio del origen de mutaciones causantes de diversos padecimientos. De este modo, podemos decir que una mutación dada, presente en dos individuos aparentemente no relacionados entre sí tiene un mismo origen, si al realizar un estudio de haplotipos, el haplotipo relacionado con la mutación en uno de los individuos es igual al haplotipo relacionado con la mutación en el otro individuo. Los estudios han dado cuenta de que los SNPs (polimorfismos de nucleótido simple) se heredan en grupos que se encuentran estrechamente relacionados en el ADN, en contraste con la tesis sostenida hasta el momento, en la que se planteaba la segregación al azar debido a la recombinación genética. A este grupo de SNPs que se heredan en bloque, es a lo que hemos denominado haplotipos.
Haplotipo en genética de poblaciones
[editar]El número (loci), así como, la longitud de haplotipos compartidos entre poblaciones, es una herramienta importante en genética de poblaciones para evaluar el grado de diferenciación genética entre las poblaciones de estudio, como se ha comentado anteriormente. Un ejemplo lo podemos encontrar en un estudio reciente llevado a cabo en habitantes de la isla de Cerdeña. En ésta, los patrones de diferenciación quedan patentes al analizar la longitud de los haplotipos compartidos con distintas poblaciones del Proyecto 1000 genomas, con una menor longitud de los mismos cuanto más distantes sean las poblaciones en cuanto a su estructura genética se refiere.[2]
Humanos
[editar]Se conoce que más de 10 000 nucleótidos que se heredan en bloque, y debido a la cantidad de SNP que se conoce que hay en el genoma humano, debe de haber en este bloque muchos SNPs. Estos SNPs que están presentes en un haplotipo pueden encontrarse en la secuencia de un gen o en la de múltiples genes pero lo que está claro que el haplotipo permite determinar el contexto en el cual actúan los genes. Un estimado reciente sugiere que entre el 65-85 % del genoma humano puede estar contenido en los bloques de haplotipos.
En octubre del año 2002 comenzó un nuevo proyecto similar al Proyecto del Genoma Humano, al cual se le denominó Proyecto Internacional HapMap en el que participan 9 grupos de investigación de los 5 continentes. Su objetivo fundamental era la determinación del haplotipo a partir de los patrones genéticos que se identificaran en muestras de sangre de 200-400 personas de diferente origen étnico.
La principal implicación que ha tenido el estudio de los haplotipos es la reducción del número potencial de diferentes genotipos. Además, existe ahora la posibilidad de identificar los rasgos heredables que comprenden múltiples variantes de SNP, detectando sólo un SNP. Esto reduce el trabajo necesario para descubrir las relaciones entre una enfermedad y uno o más polimorfismos. Este trabajo se hace típicamente mediante estudios de ligamiento, donde son genotipados los grupos de pacientes con una enfermedad (ya sea agrupados en familia, o población no relacionada entre sí) y un grupo adecuado de control y en el grupo de datos obtenidos buscan correlaciones entre el genotipo y el diagnóstico. Cuando no se conoce el gen asociado, esta aproximación requiere de un volumen grande de estudios de genotipaje. Sin embargo, si un SNP está siempre presente en el contexto de un haplotipo específico, la secuencia completa de ADN puede ser inferida con un esfuerzo mínimo.
Medicina evolutiva
[editar]El haplotipo podría comenzar también a ser una herramienta de gran valor en la medicina evolutiva. Mediante análisis de variaciones específicas y patrones de herencia como los efectos de la heterocigosidad y el desequilibrio de ligamiento (una secuencia genética que es heredada como un bloque y que no puede ser disuelta mediante los eventos de recombinación natural, lo que indica una formación relativamente reciente de ese alelo) los antropólogos, genetistas poblacionales e investigadores biomédicos podrían colectar información sobre la selección natural y las adaptaciones que relacionan los patógenos antiguos y modernos.
El conocimiento básico que pueden proveer las investigaciones de medicina evolutiva y la determinación de los haplotipos será de gran valor para el desarrollo de aplicaciones farmacéuticas. Han emergido evidencias de que los haplotipos son más relevantes clínicamente que los simples genotipos de SNP, debido a que el haplotipo puede reflejar la presencia de sitios de mutaciones no identificadas adicionales, que pueden estar relacionados con la enfermedad. También los haplotipos pueden reflejar dos o más sitios de mutaciones que pueden actuar juntos para causar una enfermedad, aunque son menos dañinos cuando están presentes en cromosomas separados.
El haplotipaje puede dar como resultado una mejor comprensión de la influencia de la genética y la transmisión de la información genética por los cromosomas dentro de poblaciones específicas. Así como un mayor entendimiento del pronóstico de enfermedades, y la interrelación entre los genes que afectan el desencadenamiento fenotípico, que también puede incluir influencias ambientales.
Se espera que todo esto sea mucho más exacto que usando sólo el genotipaje de SNP debido a que la combinación específica de múltiples polimorfismos pueden tener efectos aditivos o substractivos dirigidos a un rasgo particular que no puede predecirse mediante el genotipaje simple de SNP. Desafortunadamente, el conocimiento de los haplotipos y la composición de SNP esta aún muy incipiente. Los científicos están actualmente desarrollando medios para identificar las relaciones de los SNP, y colocarlos en los mapas de haplotipos. Hasta el momento para aplicaciones como la determinación de paternidad o los estudios forenses, se han empleado técnicas de microsatélites o STR (repeticiones de fragmentos pequeños) para detectar algunos haplotipos conocidos.
Resolución del haplotipo
[editar]El genotipo de un organismo puede no definir unequívocamente su haplotipo. Por ejemplo, consideramos un organismo diploide y dos locus bi-alélicos en el mismo cromosoma tales como SNPs. El primer locus tiene alelos A y T con tres posibles genotipos AA, AT y TT, y el segundo locus tiene G y C, dando de nuevo tres posibles genotipos GG, GC y CC. Para un individuo dado, hay entonces nueve configuraciones posibles para el genotipo en estos dos locus, tal como se muestra en el recuadro, el cual muestra los posibles genotipos que un individuo puede portar y los correspondientes haplotipos que estos determinan. Para individuos que son homocigotos para uno o ambos loci, está claro qué haplotipos son, es solo cuando un individuo es heterocigoto en ambos loci cuando la fase es ambigua.
| AA | AT | TT | |
|---|---|---|---|
| GG | AG AG | AG TG | TG TG |
| GC | AG AC | AG TC o AC TG |
TG TC |
| CC | AC AC | AC TC | TC TC |
El único método inequívoco de resolver la ambigüedad de fase es por secuenciación. Sin embargo, es posible calcular la probabilidad de un haplotipo particular cuando la fase es ambigua usando una muestra de personas individuales.
Teniendo en cuenta los genotipos para varios individuos, los haplotipos pueden ser deducidos por resolución de haplotipo o técnicas de ajuste de fase de haplotipo. Estos métodos funcionan aplicando la observación de que ciertos haplotipos son comunes en ciertas regiones genómicas. Por tanto, dado un conjunto de soluciones de haplotipos posibles, estos métodos escogen a aquellos que emplean haplotipos menos diferentes en general. Los detalles concretos de estos métodos varían - algunos están basados en los enfoques combinatorios (por ejemplo: el principio de parsimonia), mientras que otros emplean funciones de probabilidad basadas en diferentes modelos y suposiciones como la ley de Hardy-Weinberg, el modelo de teoría coalescente, o filogenia perfecta. Estos modelos son combinados con algoritmos de optimización tales como EM (expectation maximization) o MCMC (Markov Chain Monte Carlo).
Haplotipos de ADN-Y de pruebas de ADN genealógico
[editar]A diferencia de otros cromosomas, el cromosoma Y no está presente en pares. En mamíferos, cada macho tiene una única copia de este cromosoma. Esto quiere decir que no hay ninguna aleatoriedad de qué copia heredar, y tampoco (para gran parte del cromosoma) intercambio entre copias por recombinación; por eso, a diferencia de los haplotipos autosómicos, no hay efectivamente variabilidad del haplotipo del cromosoma Y entre generaciones, y un macho debe compartir el mismo cromosoma Y que su padre en gran parte, sujeto a algunas mutaciones.
En particular, el ADN-Y que aparece en los resultados numerados de una prueba de ADN-Y genealógico debe combinar, sin que aparezcan mutaciones. Dentro de la discusión genealógica y popular, esto se considera la "Firma de ADN" de un macho en particular, o de su linaje paternal.
Resultados de UEP (resultados de SNP)
[editar]Los resultados que muestran el haplotipo completo del ADN-Y a partir de la prueba de ADN del cromosoma Y pueden ser divididos en dos partes: los resultados para polimorfismos de único evento (UEPs), a veces llamados como resultados de SNP, puesto que la mayoría de los UEPs son polimorfismos de nucleótido simple, y los resultados para microsatélites de cortas secuencias repetitivas en tándem (STRs-Y), a menudo designados por números DYS.
Los resultados de UEP reflejan la herencia de los eventos que se cree que pueden ser considerados como que han ocurrido una única vez en toda la historia humana. Estos pueden ser empleados para identificar directamente el haplogrupo de ADN-Y del individuo, su lugar directamente sobre el árbol genealógico de la humanidad completa. Los diferentes haplogrupos de ADN-Y identifican a poblaciones genéticas que se encuentran a menudo localizadas geográficamente de forma concreta, reflejando las migraciones de los antepasados de la línea paterna directa de los individuos durante miles de años.
Haplotipos de STR-Y
[editar]La otra parte posible de los resultados genéticos es el haplotipo de STR-Y, el conjunto de resultados a partir de los marcadores de STR-Y evaluados.
A diferencia de los UEPs, los STRs-Y mutan mucho más fácilmente, lo cual proporciona mucha más resolución para distinguir genealogía reciente. Pero también quiere decir que, en vez de la población de descendentes de un evento genético que comparte el mismo resultado, los haplotipos de STR-Y parecen haberse dispersado, formando un grupo de resultados más o menos similares. Típicamente, este grupo tendrá un centro concreto con una mayor probabilidad, el haplotipo modal (presumiblemente cercano al haplotipo del evento original), y también una diversidad de haplotipo - el grado en el que ha llegado extenderse. Cuanto más antiguo sea el evento producido y mayor el crecimiento de la población, mayor será la diversidad de haplotipo para los individuos descendientes. Por otro lado, si la diversidad de haplotipo es más pequeña para un número en particular de descendientes, esto puede demostrar un antepasado común más reciente, o que una expansión de población ha ocurrido más recientemente.
Es importante hacer notar que, a diferencia de para UEPs, no hay garantía de que dos individuos con uno haplotipo de STR-Y similar compartan una ascendencia similar necesariamente. No hay singularidad sobre los eventos de STR-Y. Por tanto, los grupos de resultados de haplotipo de STR-Y que proceden de eventos diferentes e historias diferentes tienden a solaparse.
Así, aunque a veces un haplotipo de STR-Y podría ser directamente indicativo de un haplogrupo de ADN-Y en particular, en la mayoría de los casos es mucho el tiempo desde que se definieron los eventos de los haplogrupos, así que típicamente el grupo de resultados de haplotipo de STR-Y está relacionado con descendientes de ese evento que parece ser bastante extenso, y tiende a solaparse significativamente con los grupos (extensos de forma semejante) de haplotipos de STR-Y relacionados con otros haplogrupos, haciendo imposible pronosticar con certeza absoluta a qué haplogrupo de ADN-Y señalaría un haplotipo de STR-Y. A partir de los STRs-Y, si los UEPs no se encuentran evaluados, se pueden pronosticar las probabilidades para el origen del haplogrupo, pero no la certeza.
Un grupo de haplotipos de STR-Y similares puede indicar un antepasado común compartido, con un haplotipo modal identificable, pero solo si el grupo es suficientemente distinto del caso de individuos diferentes históricamente habiendo adoptado el mismo nombre por separado. Esto podría requerir el typing de un amplio abanico de haplotipos, para lo cual se ha propuesto ampliar el conjunto de marcadores de ADN – 24, luego 37, después 67 y quizás pronto aún más.
El objetivo del Proyecto Internacional HapMap es crear un mapa de haplotipos del genoma humano. Su intención es que este mapa sea público para así facilitar la investigación y el desarrollo de fármacos contra diversas enfermedades. Participan científicos de Canadá, China, Japón, Nigeria, Reino Unido y Estados Unidos que estudian muestras poblacionales de Yoruba (Nigeria), Tokio (Japón), Pekín (China) y Estados Unidos (personas con ancestros europeos) con frecuencias de haplotipos suficientemente diferentes como para garantizar un análisis a gran escala de cada población. Comenzó oficialmente el 27 de octubre de 2002, y se encuentra ya en su tercera fase.
Véase también
[editar]Software
[editar]El siguiente software está disponible para estimar haplotipos:
El siguiente software está disponible para evaluar haplotipos asociados con enfermedades:
Bibliografía
[editar]- ↑ http://gndp.cigb.edu.cu/Combinatoria%20Molecular/PDF/chapter18.pdf (enlace roto disponible en Internet Archive; véase el historial, la primera versión y la última).
- ↑ Genome sequencing elucidates Sardinian genetic architecture and augments association analyses for lipid and blood inflammatory markers (enlace roto disponible en Internet Archive; véase el historial, la primera versión y la última).
- ↑ «International HapMap Project».
- ↑ Purcell S., Daly M. J., Sham P. C. (2007). «WHAP: haplotype-based association analysis.». Bioinformatics 23: 255-256.
