Biossíntese
Biossíntese é um fenômeno, um processo de múltiplos passos, em que são produzidos compostos químicos complexos nos organismos vivos, como proteínas, lipídios e ácidos nucleicos a partir de reagentes mais simples, geralmente catalisados por enzimas, na qual os sustratos se convertem em produtos mais complexos.[1]
Ao contrário da síntese química, a biossíntese ocorre normalmente dentro dos organismos vivos e é uma parte vital do metabolismo. No entanto, a biotecnologia permite a realização de biossíntese in vitro.
A biossíntese relaciona-se com processos anabólicos.
Na biossíntese, os compostos simples se modificam, se convertem em outros compostos ou se unem para formar macromoléculas. Este processo geralmente consiste em vias metabólicas. Algumas dessas vias biossintéticas estão localizadas dentro de uma única organela celular, enquanto outras envolvem enzimas que estão localizadas dentro de múltiplas organelas celulares. Exemplos dessas vias biossintéticas incluem a produção de componentes de membrana lipídica e nucleotídeos. Assim, a biossíntese geralmente é sinônimo de anabolismo.
Os elementos necessários para a biossíntese incluem: compostos precursores, energia química (por exemplo, ATP), e enzimas catalíticas que podem requerer coenzimas (por exemplo, NADH, NADPH). Estes elementos criam monômeros, os blocos de construção para macromoléculas. Algumas macromoléculas biológicas importantes incluem: proteínas, que são compostas por monômeros de aminoácidos unidos por ligações peptídicas, e moléculas de ADN, que são compostas por nucleótidos unidos por ligações fosfodiéster.
Propriedades das reações químicas
[editar | editar código-fonte]A biossíntese se produz devido a uma série de reações químicas. Para que estas reações tenham lugar, são necessários os seguintes elementos:[2]
- Compostos precursores: estes compostos são as moléculas ou substratos de partida em uma reação. Estes também podem ser vistos como os reativos em um dado processo químico.
- Energia química: a energia química pode ser encontrada na forma de moléculas de alta energia. Essas moléculas são necessárias para reações energeticamente desfavoráveis. Além da hidrólise desses compostos leva a uma reação direta. Moléculas de alta energia, como o ATP, têm três fosfatos. Frequentemente, o fosfato terminal é removido durante a hidrólise e transferido para outra molécula.
- Enzimas catalíticas: estas moléculas são proteínas especiais que catalisam uma reação ao aumentar a velocidade da reação e diminuir a energia de ativação.
- Coenzimas ou cofatores: os cofatores são moléculas que ajudam nas reações químicas. Estes podem ser íons metálicos, derivados vitamínicos como NADH e acetil CoA, ou derivados não vitamínicos como ATP. No caso de NADH, a molécula transfere um hidrogênio, enquanto que a acetil CoA transfere um grupo acetilo, e o ATP transfere um fosfato.
No sentido mais simples, as reações que ocorrem na biossíntese tem o seguinte formato:[3]
Algumas variações desta equação básica que serão discutidas mais tarde com mais detalhes são:[4]
- Compostos simples que são convertidos em outros compostos, geralmente como parte de uma via de reação em várias etapas. Dois exemplos desse tipo de reação ocorrem durante a formação de ácidos nucleicos e a carga de ARNt antes da tradução. Para algumas dessas etapas, é necessária energia química:
- Compostos simples que são convertidos em outros compostos com a ajuda de cofatores. Por exemplo, a síntese de fosfolípidos requer acetil CoA, enquanto que a síntese de outro componente da membrana, os esfingolípidos, requer NADH e FADH para a formação do esqueleto da esfingosina. A equação geral para estes exemplos é:
- Compostos simples que se unem para criar uma macromolécula. Por exemplo, os ácidos graxos se unem para formar fosfolípidos. Por sua vez, os fosfolípidos e o colesterol interatuam de maneira não covalente para formar a bicapa lipídica. Esta reação pode ser descrita como segue:
Lipídios
[editar | editar código-fonte]
Muitas macromoléculas intrincadas são sintetizadas em um padrão de estruturas simples e repetidas.[5] Por exemplo, as estruturas mais simples de lipídios são os ácidos graxos. Os ácidos graxos são derivados de hidrocarbonetos ; contém uma "cabeça" de grupo carboxilo e uma "cauda" de cadeia de hidrocarboneto.[5] Esses ácidos graxos criam componentes maiores, que por sua vez incorporam interações não covalentes para formar a bicamada lipídica.[5] As cadeias de ácidos graxos são encontradas em dois componentes principais dos lipídios da membrana: o fosfolipídios e os esfingolipídios. Um terceiro componente principal da membrana, colesterol, não contém essas unidades de ácido graxo.[6]
Fosfolipídios
[editar | editar código-fonte]A base de todas as biomembranas consiste em uma estrutura bicamada de fosfolipídios.[7] A molécula de fosfolipídio é anfipática; contém uma cabeça polar hidrofílica e uma cauda não polar hidrofóbica.[5] As cabeças de fosfolipídios interagem entre si e com o meio aquoso, enquanto as caudas de hidrocarbonetos são orientadas ao centro, longe da água.[8] Essas últimas interações conduzem a estrutura de duas camadas que atua como uma barreira para íons e moléculas.[9]
Existem vários tipos de fosfolipídios; consequentemente, suas vias de síntese diferem. No entanto, a primeira etapa na síntese de fosfolipídios envolve a formação de fosfatidato ou diacilglicerol 3-fosfato no retículo endoplásmico e membrana mitocondrial externa.[8] A rota de síntese é apresentada abaixo:

A via começa com o glicerol 3-fosfato, que é convertido em lisofosfatidato por meio da adição de uma cadeia de ácido graxo fornecida pela acil coenzima A.[10] O lisofosfatidato é então convertido em fosfatidato por meio da adição de outra cadeia de ácido graxo fornecida por um segundo acil CoA; Todas essas etapas são catalisadas pela enzima glicerol fosfato aciltransferase.[10] A síntese de fosfolipídios continua no retículo endoplasmático, e a via de biossíntese difere dependendo dos componentes do fosfolipídio particular.[10]
Esfingolipídios
[editar | editar código-fonte]Como os fosfolipídios, esses derivados de ácidos graxos têm cabeça polar e cauda apolar.[6] Ao contrário dos fosfolipídios, os esfingolipídios têm um esqueleto de esfingosina.[11] Os esfingolípidos existem nas células eucariotas e são particularmente abundantes no sistema nervoso central.[8] Por exemplo, a esfingomielina é parte da bainha de mielina das fibras nervosas.[12]
Os esfingolipídios são formados a partir de ceramidas que consistem em uma cadeia de ácido graxo ligada ao grupo amino de uma estrutura esfingosina. Essas ceramidas são sintetizadas a partir da acilação da esfingosina.[12] A via biossintética da esfingosina é apresentada abaixo:

Como a imagem indica, durante a síntese da esfingosina, palmitoil-CoA e serina sofrem uma reação de condensação que tem como resultado a formação de desidrosfingosina.[8] Este produto se reduz então para formar diidrospingosina, que se converte na esfingosina através da reação de oxidação mediante FAD.[8]
Colesterol
[editar | editar código-fonte]Este lipídio pertence a uma classe de moléculas chamadas esteróis.[6] O esteróis tem quatro anéis fundidos e um grupo hidroxilo.[6] O colesterol é uma molécula particularmente importante. Não serve apenas como um componente das membranas lipídicas, mas também é um precursor de vários hormônios esteróides , como o cortisol, a testosterona e o estrogênio.[13]
O colesterol é sintetizado a partir de acetil CoA.[13] O caminho é mostrado abaixo:
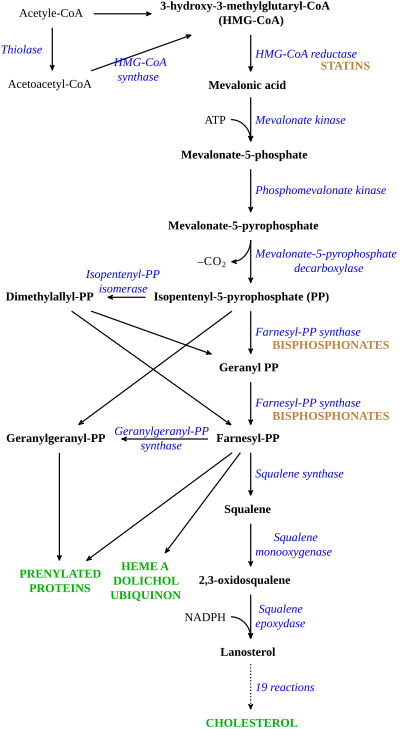
De maneira mais geral, esta síntese ocorre em três etapas, a primera etapa tem lugar no citoplasma e a segunda e terceira etapas ocorrem no retículo endoplasmático.[10] As etapas são as seguintes:[13]
- A síntese de pirofosfato de isopentenilo, o "bloco de construção" do colesterol
- A formação de esqualeno através da condensação de seis moléculas de fosfato de isopentenilo.
- A conversão de esqualeno em colesterol através de várias reações enzimáticas.
Nucleotídeos
[editar | editar código-fonte]A biosíntese de nucleotídeos implica reações catalisadas por enzimas que convertem os substratos em produtos mais complexos.[2] Os nucleotídeos são os componentes básicos do ADN e o ARN. Os nucleotídeos são compostos por um anel de cinco membros formado por açúcar de ribose no ARN e açúcar de desoxirribose no ADN; estes açúcares estão vinculados a uma base de purina ou pirimidina com uma ligação glicosídica e um grupo fosfato na posição 5 ' do açúcar.[14]
Nucleotídeos de purina
[editar | editar código-fonte]
Os nucleotídeos de ADN adenosina e guanosina consistem em uma base de purina ligada a um açúcar ribose com uma ligação glicosídica. No caso dos nucleotídeos de RNA desoxiadenosina e desoxiguanosina, as bases de purina se unem a um açúcar de desoxirribose com uma ligação glicosídica. As bases purinas nos nucleotídeos de ADN e ARN são sintetizadas em um mecanismo de reação de doze passos presente na maioria dos organismos unicelulares. Os eucariotas superiores empregam um mecanismo de reação similar em dez passos de reação. As bases purina são sintetizadas convertendo o pirofosfato de fosforibosilo (PRPP) em monofosfato de inosina (IMP), que é o primeiro intermediário chave na biossíntese da base purina.[15] A modificação enzimática adicional de IMP produz as bases de adenosina e guanosina de nucleotídeo.
- O primeiro passo na biossíntese de purinas é uma reação de condensação, realizada pela glutamina-PRPP amidotransferase. Esta enzima transfere o grupo amino da glutamina à PRPP, formando 5-fosforribosilamina. A próxima etapa requer a ativação da glicina mediante a adição de um grupo fosfato de ATP.
- GAR sintetase[16] realiza a condensação de glicina ativada en PRPP, formando ribonucleotídeo de glicinamida (GAR).
- GAR transformilase agrega um grupo formilo ao grupo amino de GAR, formando ribonucleotídeo formilglicinamida (FGAR).
- A amidotransferase[17] FGAR catalisa a adição de um grupo de nitrogênio a FGAR, formando ribonucleotídeo formilglicinamidina (FGAM).
- A FGAM ciclase catalisa o fechamento do anel, o que implica na eliminação de uma molécula de água, formando o anel de imidazol de 5 membros 5-aminoimidazol ribotida (AIR).
- A N5-CAIR sintetase transfere um grupo carboxilo, formando o intermediário N5-carboxiaminoimidazol ribonucleótido (N5-CAIR).[18]
- A N5-CAIR mutase reorganiza o grupo funcional carboxilo e o transfere o anel de imidazol, formando ribonucleotídeo carboxiaminoimidazol (CAIR). O mecanismo de dois passos da formação de CAIR a partir do AIRE se encontra principalmente em organismos unicelulares. Os eucariotas superiores possuem a enzima AIR carboxilase,[19] que transfere um grupo carboxilo diretamente ao anel de imidazol de AIR, formando CAIR.
- A SAICAR sintetase forma uma ligação peptídica entre o aspartato e o grupo carboxilo agregado do anel imidazol, formando N-succinil-5-aminoimidazol-4-carboxamida ribonucleotídeo (SAICAR).
- SAICAR liase elimina o esqueleto de carbono do aspartato agregado, deixando o grupo amino e formando ribonucleótido 5-aminoimidazol-4-carboxamida (AICAR).
- AICAR transformilase transfere um grupo carbonilo a AICAR, formando N-formilaminoimidazol-4-carboxamida ribonucleotídeo (FAICAR).
- O último passo consiste na enzima IMP sintase, que realiza o fechamento do anel de purina e forma o intermediário monofosfato de inosina.[6]
Nucleotídeos de pirimidina
[editar | editar código-fonte]
Outras bases de nucleotídeos de ADN e ARN que estão vinculadas ao açúcar da ribose através de um ligação glicosídica são a timina, a citosina e o uracilo (que só se encontra no ARN). A biosíntese de monofosfato de uridina implica uma enzima que se encontra na membrana mitocondrial interna e enzimas multifuncionais que se encontram no citosol.[20]
- O primeiro passo envolve a enzima fosfato de carbamoílo sintase na combinação de glutamina com CO2 em uma reação dependente de ATP para formar fosfato de carbamoílo.
- A aspartato carbamoiltransferase condensa o carbamoilfosfato com o aspartato para formar uridosuccinato.
- A diidroorotase realiza o fechamento do anel, uma reação que perde água, para formar diidroorotato.
- A diidroorotato desidrogenase, localizado dentro da membrana mitocondrial interna,[20] oxida o diidroorotato a orotato.
- A orotato de fosforribosilo hidrolase (OMP pirofosforilase) condensa o orotato com PRPP para formar orotidina-5'-fosfato .
- A OMP decarboxilase catalisa la conversão de orotidina-5'-fosfato em UMP.[21]
Depois que a base do nucleotídeo da uridina é sintetizada, as outras bases, citosina e timina, são sintetizadas. A biossíntese de citosina é uma reação em duas etapas envolvendo a conversão de UMP em UTP. A adição de fosfato a UMP é catalisada por uma enzima quinase. A enzima CTP sintase catalisa a seguinte etapa de reação: a conversão de UTP em CTP mediante a transferência de um grupo amino de glutamina a uridina; Este forma a base de citosina de CTP.[22] O mecanismo, que descreve a reação UTP + ATP + glutamina ⇔ CTP + ADP + glutamato, é apresentado a seguir:


A citosina é um nucleotídeo que está presente tanto no ADN como no ARN. No entanto, a uracila é encontrada apenas no RNA. Portanto, após o UTP ser sintetizado, ele deve ser convertido em uma forma deoxi para incorporar-se no ADN. Esta conversão implica a enzima ribonucleosídeo-trifosfato redutase. Esta reação que elimina o 2'-OH do açúcar ribose para gerar desoxirribose não se ve afetada pelas bases unidas ao açúcar. Esta não especificidadepermite que a ribonucleosídeo trifosfato reductase converta todos os nucleosídeos trifosfatos a desoxirribonucleotídeo mediante um mecanismo similar.[22]
Em contraste com a uracila, as bases da timina são encontradas principalmente no DNA, não no RNA. As células normalmente não contêm bases de timina que são ligadas aos açúcares ribose no RNA, indicando que as células sintetizam apenas timina ligada à desoxirribose. A enzima timidilato sintetase é responsável por sintetizar os resíduos de timina de dUMP a dTMP. Esta reação transfere um grupo metilo à base de uracilo de dUMP para gerar dTMP.[22] A reação da timidilato sintase, dUMP + 5,10-metilentetraidrofolato ⇔ dTMP + di-hidrofolato, é apresentada à direita.
ADN
[editar | editar código-fonte]
Embora existam diferenças entre a síntese de ADN eucarióticos e procarióticos , a seção a seguir denota as principais características da replicação do ADN compartilhada por ambos organismos.
O ADN é composto de nucleotídeos que são unidos por ligações fosfodiéster.[5] A síntese de ADN, que tem lugar no núcleo, é um processo semiconservativo, o que significa que a molécula de ADN resultante contém uma cadeia original da estrutura principal e uma nova cadeia.[23] A síntese de ADN é catalisada por uma família de ADN polimerases que requerem quatro trifosfatos de desoxinucleosídeos, uma cadeia de modelo e um iniciador com um 3'OH livre no qual se incorporam nucleotídeos.[24]
Para que a replicação do DNA ocorra, se cria uma bifurcação de replicação mediante enzimas chamadas helicases que desenrolam a hélice do ADN.[24] As topoisomerases na bifurcação de replicação, eliminem os ADN superenrolamentos causados pelo desenrolamento de DNA, e as proteínas de ligação a ADN de fita simples mantém os dois modelos de ADN de fita simples estabilizados antes da replicação.[14]
A síntese de ADN inicia com a ARN polimerase primase, que produz um primer de ARN com um 3'OH livre.[24] Este primer está unido ao molde de ADN de fita simples e ADN polimerase alonga a fita incorporando nucleotídeos; a ADN polimerase também corrige a fita de ANA recém-sintetizada.[24]
Durante a reação de polimerização catalisada pela ADN polimerase, é produzido um ataque nucleofílico pelo 3'OH da cadeia em crescimento no átomo de fósforo mais interno de um trifosfato de deoxinucleosídeo; esteo produz a formação de uma ponte de fosfodiéster que une um novo nucleotídeo e libera pirofosfato.[10]
Dois tipos de cadeias são criadas simultaneamente durante a replicação: a cadeia principal, que é sintetizada continuamente e cresce até a bifurcação de replicação, e a cadeia atrasada, que é realizada de forma descontínua nos fragmentos de Okazaki e longe da bifurcação de replicação.[23] Os fragmentos de Okazaki se unem covalentemente pela ADN ligase para formar uma cadeia contínua.[23] Em seguida, para completar a replicação do DNA, os primers de RNA são removidos e as lacunas resultantes são substituídas por DNA e unidas através da ADN ligase.[23]
Aminoácidos
[editar | editar código-fonte]Uma proteína é um polímero feito de aminoácidos que estão unidos por ligações peptídicas . Há mais de 300 aminoácidos encontrados na natureza dos quais somente vinte, conhecidos como os aminoácidos essenciais, são os componentes básicos das proteínas.[25] Somente as plantas verdes e a maioria dos micróbios são capazes de sintetizar todos os 20 aminoácidos essenciais que são necessários para todas as espécies vivas. Os mamíferos só podem sintetizar dez dos vinte aminoácidos essenciais. Os outros aminoácidos, valina, metionina, leucina, isoleucina, fenilalanina, lisina, treonina e triptofano para adultos e histidina e arginina para bebês são obtidos através da dieta.[26]
Estrutura básica do aminoácido
[editar | editar código-fonte]
A estrutura geral dos aminoácidos essenciais inclui um grupo amino primário, um grupo carboxilo e o grupo funcional unido ao carbono α. Os diferentes aminoácidos se identificam pelo grupo funcional. Como resultado dos três grupos diferentes unidos ao carbono α, os aminoácidos são moléculas assimétricas. Para todos os aminoácidos essenciais, exceto a glicina, o carbono α é um centro quiral. No caso da glicina, o carbono α tem dois átomos de hidrogênio, o que agrega simetria a esta molécula. Com a exceção da prolina, todos los aminoácidos encontrados na vida tem a conformação L-isoforma. A prolina tem um grupo funcional no carbono α que forma um anel com o grupo amino.[25]
Fontes de nitrogênio
[editar | editar código-fonte]
Uma etapa importante na biossíntese de aminoácidos é incorporar um grupo de nitrogênio no carbono α. Nas células, existem duas maneiras principais de incorporar grupos de nitrogênio. Uma via envolve a enzima glutamina oxoglutarato aminotransferase (GOGAT) que elimina o grupo amino amida da glutamina e a transfere a 2-oxoglutarato , produzindo dos moléculas de glutamato. Nesta reação de catálise, a glutamina serve como fonte de nitrogênio. Uma imagem que ilustra esta reação se encontra à direita.
A outra forma de incorporar nitrogênio no carbono α dos aminoácidos é a enzima glutamato desidrogenase (GDH). GDH pode transferir amônia a 2-oxoglutarato e formar glutamato. Além disso, a enzima glutamina sintase (GS) é capaz de transferir amônia a glutamato e sintetizar glutamina, que repõe glutamina.[27]
A família do glutamato de aminoácidos
[editar | editar código-fonte]A família de aminoácidos do glutamato inclui os aminoácidos que se derivam do aminoácido glutamato. Esta familia inclui: glutamato, glutamina, prolina e arginina. Esta família também inclui o aminoácido lisina, que deriva de α-cetoglutarato.[28]
A biossíntese de glutamato à glutamina é uma etapa chave na assimilação do nitrogênio discutida anteriormente. As enzimas GOGAT e GDH catalisam as reações de assimilação do nitrogênio.
Nas bactérias, a enzima glutamato 5-quinase inicia a biossíntese da prolina pela transferência de um grupo fosfato do ATP para o glutamato. A seguinte reação é catalisada pela enzima pirrolina-5-carboxilato sintase (P5CS), que catalisa a redução do grupo ϒ-carboxilo de L-glutamato 5-fosfato. Isto resulta na formação de glutamato semialdeído, que cicla espontaneamente a pirrolina-5-carboxilato. A pirrolina-5-carboxilato se reduz adicionalmente pela enzima pirrolina-5-carboxilato redutase (P5CR) para produzir um aminoácido prolina.[29]
Na primeira etapa da biossíntese de arginina em bactérias, o glutamato é acetilado transferindo o grupo acetilo de acetil-CoA à posição N-α; Isto evita a ciclização espontânea. A enzima N-acetilglutamato sintase (glutamato N-acetiltransferase) é responsável por catalisar a etapa de acetilação. Os passos subsequentes são catalisados pelas enzimas N-acetilglutamato quinase, N-acetil-gama-glutamil-fosfato redutase e acetilornitina / succinildiamino pimelato aminotransferase e produzem a N-acetil-L-ornitina. O grupo acetilo da acetilornitina é eliminado com a enzima acetilornitinase (AO) ou a ornitina acetiltransferase (OAT), e isto produz a ornitina. Então as enzimas citrulina e argininosuccinato convertem a ornitina em arginina.[30]

Existem duas vias distintas de biossíntese de lisina: a via do ácido diaminopimélico e a via do α-aminoadipato. A mais comum das duas vias sintéticas é a via do ácido diaminopimélico; consiste em várias reações enzimáticas que agregam grupos de carbono ao aspartato para produzir lisina:[31]
- A aspartato quinase inicia a rota do ácido diaminopimélico mediante a fosforilação do aspartato e a produção de fosfato de aspartilo.
- Aspartato semialdeído desidrogenase catalisa a redução dependente de NADPH do fosfato de aspartilo para produzir aspartato semialdeído.
- A 4-hidroxi-tetraidrodipicolinato sintase agrega um grupo piruvato ao β-aspartil-4-semialdeído, e é eliminada uma molécula de água. Isto provoca a ciclização e dá lugar a (2S, 4S) -4-hidroxi-2,3,4,5-tetraidrodipicolinato.
- A 4-hidroxi-tetraidrodipicolinato redutase catalisa a redução de (2S, 4S) -4-hidroxi-2,3,4,5-tetraidrodipicolinato por NADPH para produzir Δ'-piperidina-2,6-dicarboxilato (2,3,4, 5-tetraidrodipicolinato) e H2O.
- A tetraidrodipicolinato aciltransferase catalisa a reação de acetilação que produz a abertura do anel e produz N-acetil α-amino-ε-cetopimelato.
- A N-succinil-α-amino-ε-cetopimelato-glutamato aminotransaminase catalisa a reação de transaminação que elimina o grupo ceto de N-acetil α-amino-ε-cetopimelato e a substitui por um grupo amino para produzir N-succinil-L-diaminopimelato .[32]
- A N-acildiaminopimelato ciclase catalisa a desacilação de N-succinil-L-diaminopimelato para produzir L, L-diaminopimelato.[33]
- A epimerase DAP catalisa a conversão de L, L-diaminopimelato à forma meso de L, L-diaminopimelato.[34]
- A descarboxilase DAP catalisa a eliminação do grupo carboxilo, produzindo L-lisina.
A família serina dos aminoácidos.
[editar | editar código-fonte]A família serina de aminoácidos inclui: serina, cisteína e glicina. A maioria dos microorganismos e plantas obtêm o enxofre para sintetizar a metionina a partir do aminoácido cisteína. Além disso, a conversão de serina em glicina fornece os carbonos necessários para a biossíntese de metionina e da histidina.[28] Durante a biossíntese de serina,[35] a enzima fosfoglicerato desidrogenase catalisa a reação inicial que oxida o 3-fosfo-D-glicerato para produzir 3-fosfonooxipiruvato .[36] A próxima reação é catalisada pela enzima fosfoserina aminotransferase, que transfere um grupo amino do glutamato para 3-fosfonooxipiruvato para produzir L-fosfoserina.[37] A etapa final é catalisada pela enzima fosfoserina fosfatase, que desfosforila L-fosfoserina para produzir L-serina.[38]
Existem duas vias conhecidas para a biossíntese de glicina. Os organismos que usam etanol e acetato como a principal fonte de carbono utilizam a via gliconeogênica para sintetizar glicina. A outra via da biossíntese de glicina é conhecida como via glicolítica. Esta via converte a serina sintetizada a partir dos intermediários da glicólise em glicina. Na via glicolítica, a enzima serina hidroximetiltransferase catalisa a clivagem da serina para produzir glicina e transfere o grupo de carbono clivado da serina a tetraidrofolato, formando 5,10-metileno-tetraidrofolato.[39]
A biossíntese de cisteína é uma reação em duas etapas que envolve a incorporação de enxofre inorgânico. Em microorganismos e plantas, a enzima serina acetiltransferase catalisa a transferência do grupo acetilo de acetil-CoA a L-serina para produzir O-acetil-L-serina .[40] A próxima etapa da reação, catalisada pela enzima O-acetil serina (tiol) liase, substitui o grupo acetilo de O-acetil-L-serina com enxofre para produzir cisteína.[41]
A família de aminoácidos do aspartato
[editar | editar código-fonte]A família de aminoácidos de aspartato inclui: treonina, lisina, metionina, isoleucina e aspartato. A lisina e a isoleucina são consideradas parte da família do aspartato, ainda que parte de sua estrutura carbonada é derivada do piruvato. No caso da metionina, o carbono metílico é derivada da serina e o grupo de enxofre, mas, na maioria dos organismos, é derivado da cisteína.[28]
A biossíntese de aspartato é uma reação de uma etapa que é catalisada por uma única enzima. A enzima aspartato aminotransferase catalisa a transferência de um grupo amino de aspartato a α-cetoglutarato para produzir glutamato e oxaloacetato.[42]
A asparagina é sintetizada pela adição dependente de ATP de um grupo amino ao aspartato; a asparagina sintetase catalisa a adição de nitrogênio da glutamina ou amônia solúvel em aspartato para produzir asparagina.[43]

A via biossintética do ácido diaminopimélico da lisina pertence à família de aminoácidos do aspartato. Esta via envolve nove reações catalisadas por enzimas que convertem aspartato em lisina.[44]
- A aspartato quinase catalisa o passo inicial na rota do ácido diaminopimélico transferindo um fosforilo de ATP ao grupo carboxilato de aspartato, que produz aspartil-β-fosfato.[45]
- Aspartato-semialdeído desidrogenase catalisa a reação de redução por desfosforilação de aspartil-β-fosfato para produzir aspartato-β-semialdeído.[46]
- A diidrodipicolinato sintase catalisa a reação de condensação de aspartato-β-semialdeído com piruvato para produzir ácido diidrodipicolínico.[47]
- A 4-hidróxi-tetraidrodipicolinato redutase catalisa a redução do ácido diidrodipicolínico para produzir ácido tetraidrodipicolínico.[48]
- A tetraidrodipicolinato N-succiniltransferase catalisa a transferência de um grupo succinilo de succinil-CoA a ácido tetraidrodipicolínico para produzir N-succinil-L-2,6-diaminoeptanodioato.[49]
- A N-succinildiaminopimelato aminotransferase catalisa a transferência de um grupo amino do glutamato a N-succinil-L-2,6-diaminoheptanodioato para produzir ácido N-succinil-L, L-diaminopimélico.[50]
- A succinil-diaminopimelato desuccinilase catalisa a eliminação do grupo acilo do ácido N-succinil-L, L-diaminopimélico para produzir ácido L, L-diaminopimélico.[51]
- Diaminopimelato epimerase catalisa a inversão do carbono α de ácido L, L-diaminopimélico para produzir ácido meso-diaminopimélico.[52]
- A descarboxilase de siaminopimela catalisa o passo final na biossíntese da lisina que elimina o grupo de dióxido de carbono do ácido meso-diaminopimélico para produzir L-lisina.[53]
Proteínas
[editar | editar código-fonte]

A síntese de proteínas se produz através de um processo chamado tradução.[54] Durante a tradução, os ribossomas leem o material genético chamado ARNm para gerar uma cadeia polipeptídica de proteínas.[54] Este processo requer a transferência de ARN (ARNt) que serve como adaptador ao unir os aminoácidos em um extremo e interagir com o ARNm no outro extremo; o último o emparelhamento entre tRNA e mRNA garante que o aminoácido correto seja adicionado à cadeia.[54] A síntese de proteínas ocorre em três fases: iniciação, alongamento e término.[14] A tradução procariótica (arqueal e bacterial) se diferencia da tradução eucariótica; entretanto, esta seção se concentrará principalmente nas semelhanças entre os dois organismos.
Fundo adicional
[editar | editar código-fonte]Antes que a tradução possa começar, o processo de ligação de um aminoácido específico ao seu ARNt correspondente deve ocorrer. Esta reação, chamada de carregamento de ARNt, é catalisada por aminoacil-tRNA sintetase.[55] Uma tARN sintetase específica é responsável de reconhecer e carregar um aminoácido em particular.[55] Além disso, esta enzima tem regiões discriminatórias especiais para garantir a ligação correta entre o tARN e seu aminoácido relacionado.[55] O primeiro passo para unir um aminoácido a seu ARNt correspondente é a formação de aminoacil-AMP:[55]
Isso é seguido pela transferência do grupo aminoacilo de aminoacil-AMP a uma molécula de ARNt. A molécula resultante é aminoacil-ARNt:[55]
A combinação dessas duas etapas, ambas catalisadas pela aminoacil tARN sintetase, produz um tARN carregado que está pronto para adicionar aminoácidos à cadeia polipeptídica em crescimento.
Além de se ligar a um aminoácido, o ARNt tem uma unidade de três nucleotídeos chamada anticódon que se emparelha com tripletos de nucleotídeos específicos no ARNm chamado códons; os códons codificam um aminoácido específico.[56] Essa interação é possibilitada pelo ribossomo, que funciona como local para a síntese de proteínas. O ribossomo tem três sítios de ligação para ARNt: o sítio aminoacilo (sítio A), o sítio peptidilo (sítio P) e o sítio de saída (sítio E).[57]
Existem numerosos códons dentro de uma transcrição de ARNm, e é muito comum que um aminoácido seja especificado por mais de um códon; este fenômeno é chamado degeneração.[58] No total, existem 64 códons, 61 de cada código para um dos 20 aminoácidos, enquanto os códons restantes especificam a terminação da cadeia.[58]
Tradução em passos
[editar | editar código-fonte]Como mencionado anteriormente, a tradução ocorre em três fases: iniciação, alongamento e terminação.

Passo 1: Iniciação
[editar | editar código-fonte]A finalização da fase de iniciação depende dos três eventos a seguir:[14]
1.O recrutamento do ribossomo para mRNA
2. A união de um ARNt iniciador carregado no sítio P do ribossomo
3. Alinhamento correto do ribossomo com o códon inicial do ARNm
Passo 2: Alongamento
[editar | editar código-fonte]Após a iniciação, a cadeia polipeptídica é estendida através das interações anticódon: códon, e o ribossomo adiciona aminoácidos à cadeia polipeptídica um a um. As seguintes etapas devem ser realizadas para garantir a adição correta de aminoácidos:[59]
1. A união do ARNt correto no sítio A do ribossoma
2. A formação de um ligação peptídica entre o ARNt no sítio A e a cadeia polipeptídica unida ao ARNt no sítio P
3. Translocação ou avanço do complexo ARNt-ARNm por três nucleótideos
A translocação "inicia" o ARNt no sítio E e desloca o ARNt do local A para o local P, deixando o local A livre para que um ARNt de entrada adicione outro aminoácido.
Passo 3: Terminação
[editar | editar código-fonte]A última etapa da tradução ocorre quando um codón de parada ingressa no sítio A.[2] Em seguida, ocorrem as seguintes etapas:
1. O reconhecimento de códons por fatores de liberação, que causa a hidrólise da cadeia polipeptídica do ARNt localizado no sítio P[2]
2. A liberação da cadeia polipeptídica[58]
3. A dissociação e a "reciclagem" do ribossoma para futuros processos de tradução[58]
Uma tabela de resumo dos participantes chave na tradução é apresentada a seguir:
| Participantes chave na tradução | Etapa de tradução | Atividade |
|---|---|---|
| tRNA sintetase | antes da iniciação | Responsável pela carga de ARNt |
| ARNm | Iniciação, alongamento, terminação. | Modelo para síntese de proteínas; Contém regiões chamadas códons que codificam aminoácidos |
| ARNt | Iniciação, alongamento, terminação. | Liga-se aos sítios ribossômicos A, P, E; pares de bases de anticódon com o códon de mRNA para garantir que o aminoácido correto seja incorporado na cadeia polipeptídica em crescimento |
| ribossoma | Iniciação, alongamento, terminação. | Dirige a síntese de proteínas e catalisa a formação da ligação peptídica. |
Doenças associadas à deficiência de macromoléculas
[editar | editar código-fonte]
Erros nas vias biossintéticas podem ter consequências prejudiciais, como malformação de macromoléculas ou produção insuficiente de moléculas funcionais. Abaixo estão alguns exemplos que ilustram as interrupções que ocorrem devido a essas ineficiências.
- Hipercolesterolemia familiar: este transtorno se caracteriza pela ausência de receptores funcionais para as LDL.[60] As deficiências na formação do receptor de LDL podem causar receptores defeituosos que interrompem a via endocítica, inibindo a entrada de LDL no fígado e outras células.[60] Isso causa um acúmulo de LDL no plasma sanguíneo, resultando em placas ateroscleróticas que estreitam as artérias e aumentam o risco de ataques cardíacos.[60]
- Síndrome de Lesch-Nyhan: esta doença genética é caracterizada pela automutilação, deficiência mental e gota.[61] É causada pela ausência de hipoxantina-guanina fosforribosiltransferase, que é uma enzima necessária para a formação de nucleotídeos de purina.[61] A falta de enzima reduz o nível de nucleotídeos necessários e causa o acúmulo de intermediários de biossíntese, o que resulta no comportamento não usual mencionado anteriormente.[61]
- Imunodeficiência combinada grave (SCID): a SCID se caracteriza por uma perda de células T.[62] A escassez desses componentes do sistema imunológico aumenta a suscetibilidade a agentes infecciosos porque os indivíduos afetados não podem desenvolver memória imunológica.[62] Este distúrbio imunológico é o resultado de uma deficiência na atividade da adenosina deaminase, que causa uma acumulação de dATP. Estas moléculas dATP logo inibem a ribonucleotídeo reductase, o que impede a síntese de ADN.[62]
- Doença de Huntington: esta doença neurológica é causada por erros que ocurrem durante a síntese de ADN.[63] Esses erros ou mutações levam à expressão de uma proteína de huntingtina mutante, que contém resíduos de glutamina repetitivos que são codificados ao expandir as repetições de trinucleotídeos CAG no gene.[63] A doença de Huntington é caracterizada por perda neuronal e gliose. O sintomas da enfermidade incluem: transtorno do movimento, deterioração cognitiva e transtorno do comportamento[64]
Ver também
[editar | editar código-fonte]- Lipídios
- Bicapa de fosfolipídios
- Nucleotídeos
- ADN
- Replicação do ADN
- Aminoácidos proteinogênicos
- Tabela de códons
- Prostaglandina
Referências
- ↑ Brian K. Hubbard, Christopher T. Walsh: Der Aufbau von Vancomycin: so macht es die Natur. In: Angewandte Chemie. Bd. 115, Nr. 7, 2003, S. 752–789, doi:10.1002/ange.200390171
- ↑ a b c d Alberts, Bruce. Molecular biology of the cell. New York: Garland Science. ISBN 978-0815341055
- ↑ Steven S. Zumdahl, Susan A. Chemistry 8th ed. CA: Cengage Learning. ISBN 978-0547125329
- ↑ Donald Voet, Judith G. Voet, Charlotte W. Fundamentals of biochemistry : life at the molecular level 4th ed. Hoboken, NJ: Wiley. ISBN 978-0470547847
- ↑ a b c d e Harvey Lodish. Molecular cell biology 6th ed. New York: W.H. Freeman. ISBN 978-0716743668
- ↑ a b c d e David L. Nelson e Michael M. Cox. Lehninger principles of biochemistry 5th ed. New York: W.H. Freeman. ISBN 9780716771081
- ↑ Israel Hanin, Giancarlo Pepeu. Phospholipids: Biochemical, Pharmaceutical, and Analytical Considerations. [S.l.]: Springer. 318 páginas. ISBN 978-1475713664
- ↑ a b c d e Dennis E. Vance, Jean E. Vance. Biochemistry of lipids, lipoproteins and membranes 5th ed. Amsterdam: Elsevier. ISBN 978-0444532190
- ↑ J. Katsaras. Lipid bilayers : structure and interactions ; with 6 tables. Berlin [u.a.]: Springer. ISBN 978-3540675556
- ↑ a b c d e Jeremy M Berg, John L Tymoczko, and Lubert Stryer. Biochemistry 6. ed., 3. print. ed. New York: Freeman. ISBN 978-0716787242
- ↑ CR Gault, LM Obeid, YA Hannun. An Overview of sphingolipid metabolism: from synthesis to breakdown. 688, 1–23. [S.l.: s.n.] ISBN 978-1-4419-6740-4. doi:10.1007/978-1-4419-6741-1_1
- ↑ a b George J. Siegel. Basic neurochemistry : molecular, cellular and medical aspects 6th ed. Philadelphia, Pa. [u.a.]: Lippincott Williams & Wilkins. ISBN 978-0397518203
- ↑ a b c J. Robin Harris (2010). Cholesterol binding and cholesterol transport proteins : structure and function in health and disease. Dordrecht: Springer. ISBN 978-904818621 Verifique
|isbn=(ajuda) - ↑ a b c d James D. Watson. Molecular biology of the gene 6th ed. San Francisco, Calif.: Benjamin Cummings. ISBN 978-0805395921
- ↑ Kappock, TJ; Ealick, SE; Stubbe, J (outubro de 2000). «Modular evolution of the purine biosynthetic pathway.». Current Opinion in Chemical Biology. 4 (5): 567–72. PMID 11006546. doi:10.1016/s1367-5931(00)00133-2
- ↑ Sampei, G; Baba, S; Kanagawa, M; Yanai, H; Ishii, T; Kawai, H; Fukai, Y; Ebihara, A; Nakagawa, N (outubro de 2010). «Crystal structures of glycinamide ribonucleotide synthetase, PurD, from thermophilic eubacteria.». Journal of Biochemistry. 148 (4): 429–38. PMID 20716513. doi:10.1093/jb/mvq088
- ↑ Hoskins, AA; Anand, R; Ealick, SE; Stubbe, J (17 de agosto de 2004). «The formylglycinamide ribonucleotide amidotransferase complex from Bacillus subtilis: metabolite-mediated complex formation.». Biochemistry. 43 (32): 10314–27. PMID 1530153. doi:10.1021/bi049127h
- ↑ Mueller, EJ; Meyer, E; Rudolph, J; Davisson, VJ; Stubbe, J (1 de março de 1994). «N5-carboxyaminoimidazole ribonucleotide: evidence for a new intermediate and two new enzymatic activities in the de novo purine biosynthetic pathway of Escherichia coli.». Biochemistry. 33 (8): 2269–78. PMID 8117684. doi:10.1021/bi00174a038
- ↑ Firestine, SM; Poon, SW; Mueller, EJ; Stubbe, J; Davisson, VJ (4 de outubro de 1994). «Reactions catalyzed by 5-aminoimidazole ribonucleotide carboxylases from Escherichia coli and Gallus gallus: a case for divergent catalytic mechanisms.». Biochemistry. 33 (39): 11927–34. PMID 7918411. doi:10.1021/bi00205a031
- ↑ a b Srere, PA (1987). «Complexes of sequential metabolic enzymes.». Annual Review of Biochemistry. 56 (1): 89–124. PMID 2441660. doi:10.1146/annurev.bi.56.070187.000513
- ↑ Editado por Jeffrey N. Strathern, Elizabeth W. Jones, James R. Broach. The Molecular biology of the yeast Saccharomyces Cold Spring Harbor, N.Y. ed. [S.l.]: Cold Spring Harbor Laboratory. ISBN 978-0879691394
- ↑ a b c O'Donovan, GA; Neuhard, J (setembro de 1970). «Pyrimidine metabolism in microorganisms.». Bacteriological Reviews. 34 (3): 278–343. PMC 378357
 . PMID 4919542
. PMID 4919542
- ↑ a b c d Gerald Karp Geer; responsible for the revision of chapter 15 Peter van der. Cell and molecular biology : concepts and experiments 4th ed. New York: J. Wiley & Sons. ISBN 978-0471656654
- ↑ a b c d Anthony J. F. Griffiths. Modern genetic analysis 2. ed. New York: [s.n.] ISBN 978-0716731184
- ↑ a b Wu, G (maio de 2009). «Amino acids: metabolism, functions, and nutrition.». Amino Acids. 37 (1): 1–17. PMID 19301095. doi:10.1007/s00726-009-0269-0
- ↑ Mousdale, D. M.; Coggins, J. R. (1991). Amino Acid Synthesis. [S.l.: s.n.] pp. 29–56. ISBN 978-1-4899-2435-3. doi:10.1007/978-1-4899-2433-9_2
- ↑ Miflin, B. J.; Lea, P. J. (1977). «Amino Acid Metabolism». Annual Review of Plant Physiology. 28: 299–329. doi:10.1146/annurev.pp.28.060177.001503
- ↑ a b c Umbarger, HE (1978). «Amino acid biosynthesis and its regulation.». Annual Review of Biochemistry. 47 (1): 532–606. PMID 354503. doi:10.1146/annurev.bi.47.070178.002533
- ↑ Pérez-Arellano, I; Carmona-Alvarez, F; Martínez, AI; Rodríguez-Díaz, J; Cervera, J (março de 2010). «Pyrroline-5-carboxylate synthase and proline biosynthesis: from osmotolerance to rare metabolic disease.». Protein Science. 19 (3): 372–82. PMC 2866264
 . PMID 20091669. doi:10.1002/pro.340
. PMID 20091669. doi:10.1002/pro.340
- ↑ Xu, Y; Labedan, B; Glansdorff, N (março de 2007). «Surprising arginine biosynthesis: a reappraisal of the enzymology and evolution of the pathway in microorganisms.». Microbiology and Molecular Biology Reviews. 71 (1): 36–47. PMC 1847373
 . PMID 17347518. doi:10.1128/MMBR.00032-06
. PMID 17347518. doi:10.1128/MMBR.00032-06
- ↑ «MetaCyc: L-lysine biosynthesis I»
- ↑ PETERKOFSKY, B; GILVARG, C (maio de 1961). «N-Succinyl-L-diaminopimelic-glutamic transaminase.». The Journal of Biological Chemistry. 236: 1432–8. PMID 13734750
- ↑ KINDLER, SH; GILVARG, C (dezembro de 1960). «N-Succinyl-L-2,6-diaminopimelic acid deacylase.». The Journal of Biological Chemistry. 235: 3532–5. PMID 13756049
- ↑ Born, TL; Blanchard, JS (outubro de 1999). «Structure/function studies on enzymes in the diaminopimelate pathway of bacterial cell wall biosynthesis.». Current Opinion in Chemical Biology. 3 (5): 607–13. PMID 10508663. doi:10.1016/s1367-5931(99)00016-2
- ↑ «Escherichia coli K-12 substr. MG1655». serine biosynthesis. SRI International. Consultado em 12 de dezembro de 2013
- ↑ Bell, JK; Grant, GA; Banaszak, LJ (30 de março de 2004). «Multiconformational states in phosphoglycerate dehydrogenase.». Biochemistry. 43 (12): 3450–8. PMID 15035616. doi:10.1021/bi035462e
- ↑ Dubnovitsky, AP; Kapetaniou, EG; Papageorgiou, AC (janeiro de 2005). «Enzyme adaptation to alkaline pH: atomic resolution (1.08 A) structure of phosphoserine aminotransferase from Bacillus alcalophilus.». Protein Science. 14 (1): 97–110. PMC 2253317
 . PMID 15608117. doi:10.1110/ps.041029805
. PMID 15608117. doi:10.1110/ps.041029805
- ↑ Wang, W; Kim, R; Jancarik, J; Yokota, H; Kim, SH (10 de janeiro de 2001). «Crystal structure of phosphoserine phosphatase from Methanococcus jannaschii, a hyperthermophile, at 1.8 A resolution.». Structure. 9 (1): 65–71. PMID 11342136. doi:10.1016/s0969-2126(00)00558-x
- ↑ Monschau, N; Stahmann, KP; Sahm, H; McNeil, JB; Bognar, AL (1 de maio de 1997). «Identification of Saccharomyces cerevisiae GLY1 as a threonine aldolase: a key enzyme in glycine biosynthesis.». FEMS Microbiology Letters. 150 (1): 55–60. PMID 9163906. doi:10.1111/j.1574-6968.1997.tb10349.x
- ↑ Pye, VE; Tingey, AP; Robson, RL; Moody, PC (24 de setembro de 2004). «The structure and mechanism of serine acetyltransferase from Escherichia coli.». The Journal of Biological Chemistry. 279 (39): 40729–36. PMID 15231846. doi:10.1074/jbc.M403751200
- ↑ Huang, B; Vetting, MW; Roderick, SL (maio de 2005). «The active site of O-acetylserine sulfhydrylase is the anchor point for bienzyme complex formation with serine acetyltransferase.». Journal of Bacteriology. 187 (9): 3201–5. PMC 1082839
 . PMID 15838047. doi:10.1128/JB.187.9.3201-3205.2005
. PMID 15838047. doi:10.1128/JB.187.9.3201-3205.2005
- ↑ McPhalen, CA; Vincent, MG; Picot, D; Jansonius, JN; Lesk, AM; Chothia, C (5 de setembro de 1992). «Domain closure in mitochondrial aspartate aminotransferase.». Journal of Molecular Biology. 227 (1): 197–213. PMID 1522585. doi:10.1016/0022-2836(92)90691-C
- ↑ Larsen, TM; Boehlein, SK; Schuster, SM; Richards, NG; Thoden, JB; Holden, HM; Rayment, I (7 de dezembro de 1999). «Three-dimensional structure of Escherichia coli asparagine synthetase B: a short journey from substrate to product.». Biochemistry. 38 (49): 16146–57. PMID 10587437. doi:10.1021/bi9915768
- ↑ Velasco, AM; Leguina, JI; Lazcano, A (outubro de 2002). «Molecular evolution of the lysine biosynthetic pathways.». Journal of Molecular Evolution. 55 (4): 445–59. PMID 12355264. doi:10.1007/s00239-002-2340-2
- ↑ Kotaka, M; Ren, J; Lockyer, M; Hawkins, AR; Stammers, DK (20 de outubro de 2006). «Structures of R- and T-state Escherichia coli aspartokinase III. Mechanisms of the allosteric transition and inhibition by lysine.». The Journal of Biological Chemistry. 281 (42): 31544–52. PMID 16905770. doi:10.1074/jbc.M605886200
- ↑ Hadfield, A; Kryger, G; Ouyang, J; Petsko, GA; Ringe, D; Viola, R (18 de junho de 1999). «Structure of aspartate-beta-semialdehyde dehydrogenase from Escherichia coli, a key enzyme in the aspartate family of amino acid biosynthesis.». Journal of Molecular Biology. 289 (4): 991–1002. PMID 10369777. doi:10.1006/jmbi.1999.2828
- ↑ Mirwaldt, C; Korndörfer, I; Huber, R (10 de fevereiro de 1995). «The crystal structure of dihydrodipicolinate synthase from Escherichia coli at 2.5 A resolution.». Journal of Molecular Biology. 246 (1): 227–39. PMID 7853400. doi:10.1006/jmbi.1994.0078
- ↑ Cirilli, M; Zheng, R; Scapin, G; Blanchard, JS (16 de setembro de 2003). «The three-dimensional structures of the Mycobacterium tuberculosis dihydrodipicolinate reductase-NADH-2,6-PDC and -NADPH-2,6-PDC complexes. Structural and mutagenic analysis of relaxed nucleotide specificity.». Biochemistry. 42 (36): 10644–50. PMID 12962488. doi:10.1021/bi030044v
- ↑ Beaman, TW; Binder, DA; Blanchard, JS; Roderick, SL (21 de janeiro de 1997). «Three-dimensional structure of tetrahydrodipicolinate N-succinyltransferase.». Biochemistry. 36 (3): 489–94. PMID 9012664. doi:10.1021/bi962522q
- ↑ Weyand, S; Kefala, G; Weiss, MS (30 de março de 2007). «The three-dimensional structure of N-succinyldiaminopimelate aminotransferase from Mycobacterium tuberculosis.». Journal of Molecular Biology. 367 (3): 825–38. PMID 17292400. doi:10.1016/j.jmb.2007.01.023
- ↑ Nocek, BP; Gillner, DM; Fan, Y; Holz, RC; Joachimiak, A (2 de abril de 2010). «Structural basis for catalysis by the mono- and dimetalated forms of the dapE-encoded N-succinyl-L,L-diaminopimelic acid desuccinylase.». Journal of Molecular Biology. 397 (3): 617–26. PMC 2885003
 . PMID 20138056. doi:10.1016/j.jmb.2010.01.062
. PMID 20138056. doi:10.1016/j.jmb.2010.01.062
- ↑ Pillai, B; Cherney, M; Diaper, CM; Sutherland, A; Blanchard, JS; Vederas, JC; James, MN (23 de novembro de 2007). «Dynamics of catalysis revealed from the crystal structures of mutants of diaminopimelate epimerase.». Biochemical and Biophysical Research Communications. 363 (3): 547–53. PMID 17889830. doi:10.1016/j.bbrc.2007.09.012
- ↑ Gokulan, K; Rupp, B; Pavelka, MS, Jr; Jacobs, WR, Jr; Sacchettini, JC (16 de maio de 2003). «Crystal structure of Mycobacterium tuberculosis diaminopimelate decarboxylase, an essential enzyme in bacterial lysine biosynthesis.». The Journal of Biological Chemistry. 278 (20): 18588–96. PMID 12637582. doi:10.1074/jbc.M301549200
- ↑ a b c Robert F. Weaver. Molecular biology 3rd ed. Boston: McGraw-Hill Higher Education. ISBN 978-0-07-284611-9
- ↑ a b c d e Geoffrey M. Cooper (2000). The cell : a molecular approach 2nd ed. Washington (DC): ASM Press. ISBN 978-0878931064
- ↑ Jackson, R.J.; et al. (fevereiro de 2010). «The mechanism of eukaryotic translation initiation and principles of its regulation». Molecular Cell Biology. 10: 113–127
- ↑ Green, Rachel; Noller, Harry F.; et al. (1997). «Ribosomes and Translation». Annu. Rev. Biochem. 66: 679–716. PMID 9242921. doi:10.1146/annurev.biochem.66.1.679
- ↑ a b c d Pestka (editors),, Herbert Weissbach, Sidney (1977). Molecular Mechanisms of protein biosynthesis. New York: Academic Press. ISBN 978-0127442501
- ↑ Frank, J; Gao, Haixiao; et al. (setembro de 2007). «The process of mRNA–tRNA translocation». PNAS. 104 (50): 19671–19678. PMC 2148355
 . PMID 18003906. doi:10.1073/pnas.0708517104
. PMID 18003906. doi:10.1073/pnas.0708517104
- ↑ a b c Bandeali, Salman J.; Daye, Jad; Virani, Salim S. (30 de novembro de 2013). «Novel Therapies for Treating Familial Hypercholesterolemia». Current Atherosclerosis Reports. 16 (1). 382 páginas. PMID 24293346. doi:10.1007/s11883-013-0382-0
- ↑ a b c Kang, Tae Hyuk; Park, Yongjin; Bader, Joel S.; Friedmann, Theodore; Cooney, Austin John (9 de outubro de 2013). «The Housekeeping Gene Hypoxanthine Guanine Phosphoribosyltransferase (HPRT) Regulates Multiple Developmental and Metabolic Pathways of Murine Embryonic Stem Cell Neuronal Differentiation». PLoS ONE. 8 (10): e74967. PMC 3794013
 . PMID 24130677. doi:10.1371/journal.pone.0074967
. PMID 24130677. doi:10.1371/journal.pone.0074967
- ↑ a b c Ken Murphy, Paul Travers, Mark Walport. Janeway's Immunobiology 8. ed. Oxford: Taylor & Francis. ISBN 978-0815342434
- ↑ a b Hughes, Donald C. Lo, Robert E. Neurobiology of Huntington's disease: applications to drug discovery 2nd ed. Boca Raton: CRC Press/Taylor & Francis Group. ISBN 978-0849390005
- ↑ Biglan, Kevin M.; Ross, Christopher A.; Langbehn, Douglas R.; Aylward, Elizabeth H.; Stout, Julie C.; Queller, Sarah; Carlozzi, Noelle E.; Duff, Kevin; Beglinger, Leigh J. (26 de junho de 2009). «Motor abnormalities in premanifest persons with Huntington's disease: The PREDICT-HD study». Movement Disorders. 24 (12): 1763–1772. PMC 3048804
 . PMID 19562761. doi:10.1002/mds.22601
. PMID 19562761. doi:10.1002/mds.22601


![{\displaystyle {\ce {Reactante ->[][enzima] Produto}}}](/media/api/rest_v1/media/math/render/svg/89400c7a33fcafc6b400fe711dc9ab72944c80f5)

![{\displaystyle {\ce {{Mol{\acute {e}}cula~precursora}+Cofactor->[][enzima]macromol{\acute {e}}cula}}}](/media/api/rest_v1/media/math/render/svg/878fcfb41c2d0584dccf89d65b351b93ca3a1ba4)


